【python】数字を1桁ずつに分解
こういう処理がしたいときがある。
i = 2049 lst = [] while i > 0: lst.append(i%10) i //= 10 # 必須 lst.reverse() print(lst) # 結果-> [2, 0, 4, 9]
「もうできたじゃん」という意見もあると思うが、C言語のコードみたいでいかにもpythonicじゃない。
これはこれで良いとして、もっと良い方法がないか考えてみる。
先行研究
同じことを考えた人はたくさんいるようだ。日本語でググって出てきたページを幾つか貼る。
python で 数値を桁別に取得するには - スタック・オーバーフロー
数値を桁ごとにリストに格納する方法,またその逆(Python) - done is better than perfect
Python - python リスト型 変数|teratail
方法としては、上の方法の変形か、strに変換して1桁ずつ読むかのどちらかという感じ。
strに変換するというのは次のようなコード。
[int(i) for c in str(i)]
記述量は少ないが、いかんせん無駄に型変換が走るのはスマートではない。
別の方法1
再帰で書いてみた。
def digit(i, lst=[]): if i > 0: lst.append(i%10) return digit(i//10, lst) else: lst.reverse() return lst print(digit(2049)) # 結果-> [2, 0, 4, 9]
lispだったら綺麗な奴。pythonだと微妙かも。lstを使い回すのが嫌なら、下の書き方もある。
def digit(i): if i > 0: return digit(i//10) + [i%10] else: return []
どうせ末尾再帰最適化なんてされないので、listオブジェクト生成のオーバーヘッド以外にデメリットはない(そのオーバーヘッドがでかかったりするが)。appendを使えばそれすら消せそう。
そして、これができるということは、関数定義だけならワンライナーにもできる。
digit = lambda i:(digit(i//10) + [i%10]) if i > 0 else []
なぜこんなことをするのか? 私の趣味です。
別の方法2
ジェネレータを使って書くこともできると思う。やってない。
別の方法3
思いつかなかった・・・。
まとめ
普通にC言語っぽく書けば良い気がしてきたなぁ・・・。
追記
実行速度を測っていなかったので、計測してみました。
import timeit def f1(i): lst = [] while i > 0: lst.append(i%10) i //= 10 # 必須 lst.reverse() return lst def f2(i): return [int(x) for x in str(i)] print(timeit.timeit(lambda :f1(1))) print(timeit.timeit(lambda :f2(1))) """ => 0.47740281200094614 1.1206639419979183 """ print(timeit.timeit(lambda :f1(1234567890))) print(timeit.timeit(lambda :f2(1234567890))) """ => 2.2151244249980664 3.7194496169977356 """ print(timeit.timeit(lambda :f1(int("1234567890"*10)))) print(timeit.timeit(lambda :f2(int("1234567890"*10)))) """ => 28.711613783001667 25.051117279996106 """
桁数が小さければ割り算で処理する方法が速いが、大きくなるとどこかで逆転する。違いは倍程度には収まるという結果になりました。
まあ、そしたらstrに変換して1桁ずつでも良いかという気もしなくもなく・・・。
【python】in演算子は遅いのか?
はじめに
inの速度は謎である。とりあえず、なんとなく速くはないイメージはあるといえばあるので、ループの中でinを書くとちょっと罪悪感のようなものを感じる。
が、実際のところどうなのだろう?
こちらのページに計算量は出ている。
Pythonistaなら知らないと恥ずかしい計算量のはなし - Qiita
それによると、リストに対するinは平均O(n)で最悪はよくわからない。setに対するinは平均O(1)、最悪O(n)。
つまりリストに対してin演算子を使うと線形探索される。一方、setはhashで実装されていると主張している。まあ、頷ける話ではある。
とはいえ、計算量だけわかってもしょうがない。なので実測することにした。
検証
次のようなベンチマークを考えた。
- range(n)の値を持つコレクションを作り、range(2*n)の値に対してin演算子を実行。トータルの実行時間を測る。
対象にしたコレクション型はlist, set, dict(dictのkey)の3通り。nは100, 200, 400, 600, 800, 1000, 2000, 4000, 6000, 8000, 10000。CPUはintel i7 3540Mで、プログラムはwindows上で動くvmware playerの仮想環境上のubuntuで実行。python 3.5.1。
コードを以下に示す。
# coding: UTF-8 import time import pandas as pd import matplotlib.pyplot as plt def measure(n, mode): if mode == "list": collection = list(range(n)) elif mode == "set": collection = set(range(n)) elif mode == "dkey": collection = dict(zip(range(n), range(n))) else: return None # measure overhead of for t1 = time.time() for i in range(2*n): pass t2 = time.time() overhead = t2 - t1 t1 = time.time() for i in range(2*n): i in collection t2 = time.time() return t2 - t1 - overhead def main(): df = pd.DataFrame([], columns=["n", "list_time", "set_time", "dict_key_time"]) for n in [100, 200, 400, 600, 800, 1000, 2000, 4000, 6000, 8000, 10000]: list_time = measure(n, "list") set_time = measure(n, "set") dkey_time = measure(n, "dkey") s = pd.Series([n, list_time, set_time, dkey_time], index=df.columns) df = df.append(s, ignore_index=True) print(df) df.plot.line(x=["n"]) plt.savefig("result.png") plt.yscale('log') plt.savefig("result_log.png") if __name__ == "__main__": main()
結果
プログラムのテキスト出力を以下に示す。
n list_time set_time dict_key_time 0 100.0 0.000230 0.000004 0.000004 1 200.0 0.000949 0.000008 0.000008 2 400.0 0.003813 0.000044 0.000018 3 600.0 0.008644 0.000030 0.000031 4 800.0 0.015412 0.000040 0.000040 5 1000.0 0.024412 0.000055 0.000068 6 2000.0 0.097047 0.000107 0.000109 7 4000.0 0.402630 0.000255 0.000235 8 6000.0 0.882451 0.000401 0.000443 9 8000.0 1.565103 0.000492 0.000473 10 10000.0 2.448165 0.000576 0.000587
結果のグラフを次の図に示す。

とりあえず、listが劇的に遅い。しかも、リストの大きさに比例するどころか、二次関数みたいな感じになっている(縦軸logの図を見る限り指数関数よりはマシ)。CPUキャッシュなどの影響だと思う。
set, dictのkeyは互角の速度で、listと比べるといかなる要素数においても劇的に速い。1万要素(in演算2万回)で0.000612秒。O(n)と言えるかどうかはぶっちゃけ微妙。hashは衝突回避処理があるので、それが走ると遅くなるのだろうか。それともこれもキャッシュ関係?
結論
listに対するin演算は激遅だった。これは大きいリストに対しては使いたくないと思う。
set, dictのkeyに対するin演算は割と許せる速度なので、使うならこっち。だいたいリストの1/100~1/1000の速度が期待できる。リストでinすると15分かかる処理も1~10秒程度でできるということであり、つまり素晴らしい効能がある。
- listに対してたくさん呼ぶのは地獄
- in演算は無条件で遅い訳ではない。むしろsetなどに対してはそうとう速いので、基本的に使っても大丈夫
これが結論である。なんだか、実際の数字が線形より悪かったり、組み込みのsetが優秀だったりっていうのは、少し意外な感じだった。
cross_val_scoreはもうやめようね。一発で交差検証するにはcross_validateを使う
はじめに
scikit-learnで交差検証を行い、評価指標を算出する方法としては、cross_val_scoreがよくオススメされています。実際、「sklearn 交差検証」みたいな検索キーワードでググるとこの関数がよく出てきます。しかし、この関数は複数の評価指標を算出することができず、一つのスコアしか出力してくれません。
これでどういうとき困るかというと、Accuracy, Precision, Recall, F1をすべて出したい・・・というとき、困ります。基本的にこれらはぜんぶ出して評価するものという考え方のもと検証しようとすると、うまくいかないのです。その辺りを柔軟に制御するために、これまで私は自分で交差検証のコードを書いてきました。
しかし、そんな必要はありませんでした。cross_validateという関数を使えばいいのです。
・・・と、大げさに書いてみましたが、実はこの関数はsklearnのオンラインドキュメントのAPI ReferenceのModel Validationの筆頭に出ています。
API Reference — scikit-learn 0.21.2 documentation
じゃあなんでcross_val_scoreがオススメされるの? というと、cross_validateの方が若干新しいからです。これが使えるのは0.19以降です。それだけの理由ですね。古い情報がそのまま定着してしまって、もっと良いものが出てきてもそれが広まらないというのはよくあることです。
cross_validateの方が何かと柔軟なので、こちらを使いましょう。以下では淡々と説明していきます。
スポンサーリンク
cross_validate
ドキュメントの個別ページはここです。
sklearn.model_selection.cross_validate — scikit-learn 0.21.2 documentation
こんな関数になっています。
sklearn.model_selection.cross_validate(estimator, X, y=None, groups=None, scoring=None, cv=None, n_jobs=1, verbose=0, fit_params=None, pre_dispatch=‘2*n_jobs’, return_train_score=’warn’)
あまりcross_val_scoreと代わり映えしないと言えばしないのですが、scoringに指定できるものがcross_val_scoreとは違います。ちょっと長いですが、当該部分を全文引用します。
scoring : string, callable, list/tuple, dict or None, default: None
A single string (see The scoring parameter: defining model evaluation rules) or a callable (see Defining your scoring strategy from metric functions) to evaluate the predictions on the test set.
For evaluating multiple metrics, either give a list of (unique) strings or a dict with names as keys and callables as values.
NOTE that when using custom scorers, each scorer should return a single value. Metric functions returning a list/array of values can be wrapped into multiple scorers that return one value each.
See Specifying multiple metrics for evaluation for an example.
If None, the estimator’s default scorer (if available) is used.
けっこう凄いことがかいてあります。嬉しいのはlist/tuple, dictが渡せるところです。どう嬉しいかはすぐにわかります。
実例
実際のコードを見ないと良さが伝わらないと思うので、簡単な例を示します。
# coding: UTF-8 from pprint import pprint from sklearn.datasets import load_iris from sklearn.naive_bayes import GaussianNB from sklearn.model_selection import cross_validate, StratifiedKFold def main(): iris = load_iris() gnb = GaussianNB() scoring = {"p": "precision_macro", "r": "recall_macro", "f":"f1_macro"} skf = StratifiedKFold(shuffle=True, random_state=0) scores = cross_validate(gnb, iris.data, iris.target, cv=skf, scoring=scoring) pprint(scores) if __name__ == "__main__": main()
scoresの中身がどうなってるのか? というのがミソの部分で、こうなっています。
{'fit_time': array([0.00096989, 0.00186491, 0.00081563]),
'score_time': array([0.00263953, 0.0025475 , 0.00260162]),
'test_f': array([0.9212963 , 0.98037518, 0.95816993]),
'test_p': array([0.9251462 , 0.98148148, 0.96296296]),
'test_r': array([0.92156863, 0.98039216, 0.95833333]),
'train_f': array([0.95959596, 0.95955882, 0.95096979]),
'train_p': array([0.95959596, 0.96067588, 0.95122655]),
'train_r': array([0.95959596, 0.95959596, 0.95098039])}これを見た瞬間、「もう自分で交差検証を書く必要はない」と私は思いました。欲しい情報はぜんぶ出せます。
・・・といいつつ
不満点はまったくない訳ではなく、
- 各foldでfitさせた分類器がほしい、とか
- 各foldの正解/予測ラベルを生でほしい、とか
いろいろ思うところはあるので、そういうものが必要なときは他のモジュールを探すか(把握しきれてないです、はい)、自分で書くことになります。
それでも普通に数字を出して評価する「だけ」なら一行一発で済む関数があるというのは素晴らしいことです。手間が省けます。素晴らしい。cross_val_scoreはその域には達してないと判断していましたが、cross_validateは使えそうです。
これで簡単に交差検証が書けるようになりました。
まとめ
cross_validateを使おう。
【python】複数の選択肢から確率で選ぶ
おみくじや福引きのようなもの、あるいは強化学習の実装などでタイトルのような「複数のものから確率で選ぶ」処理が必要になることがある。
これについては以前にもこのような記事を書いた。
【python】一定の確率で違う選択をする - 静かなる名辞
この方法でも上手くいくのだが、選択肢が複数になった場合が面倒くさかったり、確率を後から変えるのが大変だったりと、それなりにデメリットがあった。
理想的には、確率のリストなどを渡せばよしなに処理してくれるものがほしい。こんな風に。
何か凄いもの(確率のリスト)
# -> リストのindexを0:15%, 1:35%, 2:20%, 3, 30%の確率で返す
できないと思っていたが、numpyでできることが判明した。ただしversion 1.7.0以上が必要。
numpy.random.choice — NumPy v1.15 Manual
たとえば、a,b,c,dを[0.15, 0.35, 0.2, 0.3]の確率で選びたい場合、単にこうすれば良い。
np.random.choice(["a", "b", "c", "d"], p=[0.15, 0.35, 0.2, 0.3]) # -> "a", "b", "c", "d"のいずれかが確率で返る
想像以上に簡単だ・・・。
インデックスを返させることもできる。
np.random.choice(4, p=[0.15, 0.35, 0.2, 0.3]) # -> 0, 1, 2, 3のいずれかが確率で返る
0,1,2,3を選んでくれる。素晴らしい。
更に、複数の結果を吐かせることもできる。
np.random.choice(4, size=10, p=[0.15, 0.35, 0.2, 0.3]) # -> 一例:array([3, 2, 3, 3, 1, 3, 2, 1, 1, 0])
更に更に、replaceというオプションがある。これは重複するかどうかを指定する(ふくびきで出た玉を一々中に戻すかどうかに相当)。defaultはTrueで、玉を中に戻すことに相当する。Falseにすると次のようなことができる。
np.random.choice(5, 5, replace=False) # -> array([3, 0, 4, 1, 2])
これはランダムサンプリングするときに使えるだろう。ただし、同時にpオプションを指定すると上手く動かない。次のエラーが出る。
ValueError: Cannot take a larger sample than population when 'replace=False'
何はともあれ、こんな便利なものがあることは知らなかった。普段numpyのドキュメントを積極的に読もうと思わないので、こういうことに気づかないパターンが多い。
機会があったら使っていこう・・・。
【python】RandomTreesEmbeddingを試す(1)
RandomTreesEmbeddingはsklearnにたくさんある謎クラスの一つ*1。
たぶんスパースコーディングに決定木を使いましょうね~系の奴なんだと思う。
ドキュメントを読むと、なんとなく雰囲気はわかる。
sklearn.ensemble.RandomTreesEmbedding — scikit-learn 0.20.1 documentation
あー、なるほど、決定木を作った後、木のパスを符号にしてるのね(わかってない。でもハフマン符号みたいなものかな、という見当はつく)。
とりあえずどんなものか試してみる。というかこの記事は本当に試してみただけ。
なにはともあれirisから
いつも通り分類問題の前処理をやらせてみよう。
プログラム。
# coding: UTF-8 from sklearn.datasets import load_iris, load_digits from sklearn.ensemble import RandomTreesEmbedding as RTE,\ RandomForestClassifier as RFC from sklearn.naive_bayes import GaussianNB from sklearn.model_selection import cross_validate def test_func(dataset, clf, embed=True): if embed: rte = RTE(n_estimators=100, sparse_output=False, n_jobs=-1) X = rte.fit_transform(dataset.data) else: X = dataset.data print(X.shape) scoring = {"p": "precision_macro", "r": "recall_macro", "f":"f1_macro"} scores = cross_validate(clf, X, dataset.target, scoring=scoring, return_train_score=False, n_jobs=-1) print("p:{0:.5f} r:{1:.5f} f:{2:.5f}".format( scores["test_p"].mean(), scores["test_r"].mean(), scores["test_f"].mean())) def main(): iris = load_iris() digits = load_digits() gnb = GaussianNB() rfc = RFC(n_estimators=1000, n_jobs=-1) print("iris") test_func(iris, gnb, embed=False) test_func(iris, rfc, embed=False) test_func(iris, gnb, embed=True) test_func(iris, rfc, embed=True) print("digits") test_func(digits, gnb, embed=False) test_func(digits, rfc, embed=False) test_func(digits, gnb, embed=True) test_func(digits, rfc, embed=True) if __name__ == "__main__": main()
結果
iris (150, 4) p:0.93611 r:0.93423 f:0.93411 (150, 4) p:0.96768 r:0.96732 f:0.96731 (150, 2155) p:0.96234 r:0.96038 f:0.96027 (150, 2189) p:0.96234 r:0.96038 f:0.96027 digits (1797, 64) p:0.84221 r:0.81910 f:0.82039 (1797, 64) p:0.94671 r:0.94478 f:0.94473 (1797, 2503) p:0.90086 r:0.89582 f:0.89597 (1797, 2488) p:0.93643 r:0.93373 f:0.93403
まあ思った通り、4次元から2000次元以上に張られてしまう奴だった。ナイーブベイズだと分類精度に恩恵があるっぽいが、ランダムフォレストで効く訳もなく。
ちなみに、木の数を増やすと次元数はだいたい比例して増える。おっかない。まあ、逆に言えば木の数で適当な次元数に調整できるということでもあるのだが(他の決定木のパラメタでもできる)。
まとめ
本来の使い方じゃないと思うので、そのうちちゃんとした記事を書きます・・・。
たぶん画像処理とかの問題を解くのに使えるんだと思う。
*1:ひどい言い方だけど、「僕にとって謎」という意味である。予防線張っとく
【python】sklearnのPCAでloading(主成分負荷量)を計算する
PCA(主成分分析)のloading*1がほしいときがあります。
sklearnでは一発では出ません。
ドキュメントはここ。
sklearn.decomposition.PCA — scikit-learn 0.21.2 documentation
目次
PCA.components_は確かにあるけど・・・
結論から先に言うと、PCA.components_はノルム1の固有ベクトルです。ノルムを測ってみましょう。
>>> import numpy as np >>> from sklearn.datasets import load_iris >>> from sklearn.decomposition import PCA >>> iris = load_iris() >>> pca = PCA(n_components=2) >>> pca.fit(iris.data) PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False) >>> pca.components_ array([[ 0.36158968, -0.08226889, 0.85657211, 0.35884393], [ 0.65653988, 0.72971237, -0.1757674 , -0.07470647]]) >>> np.linalg.norm(pca.components_, axis=1) array([1., 1.])
まあ、loadingも固有ベクトルには違いないのですが、ノルムを整えてやる必要があります。
loadingを計算しよう
教科書などによく書いてあることですが、第主成分に対応する元の変数のloadingは次の式で出せます。
は固有値。 eigenvectorは固有ベクトルで、元の変数の数だけ次元がありますから、これで良いわけです(雑な説明ですが・・・)。
ということで、pythonで同様にやってみましょう。固有値はexplained_varianceに入っています。
>>> pca.components_*np.c_[np.sqrt(pca.explained_variance_)] # 縦ベクトルに変換する必要がある array([[ 0.74322652, -0.16909891, 1.76063406, 0.73758279], [ 0.32313741, 0.35915163, -0.08650963, -0.03676921]])
できました。これがloadingです。・・・と思ったけど、1を超えちゃってますね。なんてこった。
罠だった
固有値は分散なので、データのスケールに依存します。
とりあえず入力をスケーリングしてみよう。上の式は相関行列から行くときのものでした。なのでこれで平気なはず。
>>> from sklearn.preprocessing import StandardScaler as SS >>> ss = SS() >>> data = ss.fit_transform(iris.data) >>> pca.fit(data) PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False) >>> pca.components_*np.c_[np.sqrt(pca.explained_variance_)] array([[ 0.89421016, -0.45081822, 0.99500666, 0.96822861], [ 0.35854928, 0.89132754, 0.02031465, 0.06299656]])
1を超えなくなくてめでたし、ということよりも、数字が変わったことの方が問題で、これで本当に正しいのかという疑念が生じてきました。
確認のために元の特徴と主成分の相関係数を直接測ってみます。
>>> X = pca.fit_transform(data) >>> np.corrcoef(np.hstack([iris.data, X]), rowvar=False) array([[ 1.00000000e+00, -1.09369250e-01, 8.71754157e-01, 8.17953633e-01, 8.91224479e-01, 3.57352114e-01], [-1.09369250e-01, 1.00000000e+00, -4.20516096e-01, -3.56544090e-01, -4.49312976e-01, 8.88351481e-01], [ 8.71754157e-01, -4.20516096e-01, 1.00000000e+00, 9.62757097e-01, 9.91684422e-01, 2.02468206e-02], [ 8.17953633e-01, -3.56544090e-01, 9.62757097e-01, 1.00000000e+00, 9.64995787e-01, 6.27862218e-02], [ 8.91224479e-01, -4.49312976e-01, 9.91684422e-01, 9.64995787e-01, 1.00000000e+00, 2.08904471e-17], [ 3.57352114e-01, 8.88351481e-01, 2.02468206e-02, 6.27862218e-02, 2.08904471e-17, 1.00000000e+00]])
下の二行の4列目までを見てください。微妙に誤差があるような気はしますが(小数点以下3桁でずれてきてるので微妙ってほど微妙でもないが)、たぶん同じ数字になっています。
微妙な誤差については、丸め誤差などが累積した、実は計算間違ってる、といった理由が考えられます。前者ならまだ許せるけど、後者はやだな・・・。
共分散行列のときはどうするのか
どうするんだろうね・・・。
2019/06/22追記
手順は増えますが、スケールを考慮すれば同様に行えるようです。
出典:http://manabukano.brilliant-future.net/document/text-PCA.pdf p.10
loadingを使うと何が良いのか
相関係数なので、「どれくらい効いてるか」がよくわかります。よくある0.3以下なら~とか0.7以上なら~という論法ができます。それだけといえばそれだけ。
このように取扱が大変なので、固有ベクトルのまま解釈した方が楽かもという気がしてきました。各主成分の寄与率はexplained_variance_ratio_で得られる訳だし、寄与率の大きい軸の固有ベクトルの大きい次元を見ればどんな感じかはわかるし・・・。
でもまあ、一応(入力をスケーリングすれば)大体出るということはわかったので、これでよしとします。
共分散行列でやるときのやり方は、どなたか詳しい方に教えて頂けると助かります。
*1:主成分負荷量、あるいは因子負荷量とも(なぜか)言われますが、この記事ではloadingで通します。けっきょくヘタに和訳しないのがいちばんわかりやすい
【python】sklearnで因子分析を試す
pythonで因子分析をやる人はあまりいないようだが、sklearnにはしっかりモデルが存在している。ついさっき気づいた。
sklearn.decomposition.FactorAnalysis — scikit-learn 0.20.1 documentation
因子分析自体は前からどんなものなのか興味があり、かといってググるとRだったりSPSSだったりばっかり出てきて辟易していたのだが、sklearnにあると都合が良い。さっそく使ってみよう。
目次
とりあえずirisをプロットする
私だけでも何十回もやってきた、世界中では何万回とやられてきたirisの二次元可視化をやってみる。
次のようなコードを書いた。
# coding: UTF-8 from copy import deepcopy from sklearn.datasets import load_iris from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA, FactorAnalysis as FA from sklearn.pipeline import Pipeline import matplotlib.pyplot as plt def decomp_and_plot(dataset, model, file_name): X = model.fit_transform(dataset.data) plt.figure() plt.scatter(X[:,0], X[:,1], c=dataset.target/len(dataset.target_names)) plt.savefig(file_name) def main(): iris = load_iris() ss = StandardScaler() pca = PCA(n_components=2) pl = Pipeline([("scaler", ss), ("pca", deepcopy(pca))]) fa = FA(n_components=2, max_iter=5000) decomp_and_plot(iris, pca, "pca_plt.png") decomp_and_plot(iris, pl, "spca_plt.png") decomp_and_plot(iris, fa, "fa_plt.png") if __name__ == "__main__": main()
PCA、変数をスケーリングしたPCA(相関行列を使うことと等価)、因子分析でそれぞれplotしてみる。
結果はこれ。



とりあえずcomponentsを見る
次のようなmain2を作り、実行した。
def main2(): iris = load_iris() print(iris.feature_names) print("pca") pca = PCA(n_components=2) pca.fit(iris.data) print(pca.components_) print("fa") fa = FA(n_components=2, max_iter=5000) fa.fit(iris.data) print(fa.components_)
結果
['sepal length (cm)', 'sepal width (cm)', 'petal length (cm)', 'petal width (cm)'] pca [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [ 0.65653988 0.72971237 -0.1757674 -0.07470647]] fa [[ 0.72577591 -0.17754023 1.75733754 0.73196365] [-0.37036948 -0.24060118 0.02793388 0.04121372]]
プロット結果から予想される通り、両者のcomponentsはよく似通っている。
これがloadingなのかどうかはぶっちゃけよくわからないのだが(というか1を超えてくる時点でたぶん違うのだろうが)、とりあえずloadingだと思って解釈する。
第一因子は花弁の長さと幅、がく片の長さに対応しているので花の大きさに対応しているっぽい。花の大きさとがく片の幅はなぜか若干反比例する。
第二因子は花弁に関する係数が小さいので、がく片の大きさを表す因子と言って良さそうである。
こんなところか。
使えることはわかった
だから何? って言われると、正直答えに窮しますが・・・とにかく使えます。主成分分析で良いじゃんと言われたら何も言い返せません。
【python】改行せずに代入文と等価のことをする
pythonワンライナーを書く上で障害になるのは、代入文の存在である。
代入は関数ではなく文なので、素直に書くと一行を消費してしまうし、lambdaやコレクション型の中にも入れられない。
よく知られた対策としては、グローバル変数テーブルを直接書き換えるという大技がある。
>>> globals().__setitem__("hoge", 1) >>> hoge 1
でも__setitem__とか呼ぶのは、はっきり言ってキモい。それに、ローカル変数は書き換えられないという問題もある。もっと普通のpythonの枠内でpythonワンライナーを書きたい。
長らくこの問題に悩んでいたが、ついに等価の表現を発見した。リストならアンダーバー付きのメソッドを呼ばなくても行ける。
>>> l = [1] >>> (l.pop(0), l.append(2))[-1] >>> l [2]
ワンライナーとして変数辞書と同様に使える表現を考えてみる。lispのalistを参考にして作る。
(lambda setalist, getalist, alist: # 環境のalistとアクセス用関数を定義(lispのletと同様) ((setalist("hoge", 1, alist), # 以下main print(getalist("hoge", alist)), setalist("hoge", 2, alist), print(getalist("hoge", alist)), setalist("fuga", "fuga", alist), print(getalist("fuga", alist)), print(getalist("hoge", alist)), setalist("lst", [], alist), getalist("lst", alist).extend([1,2,3,4,5]), print(getalist("lst", alist)[0:2]) # mainここまで ,)))( # ここから一番外のlambdaの引数 (lambda key, value, alist: # setalistの実体 (lambda search_result: ((alist.pop(search_result[0]) if search_result != [] else None), alist.append((key, value)), value)[-1])([i for (i, (k,v)) in enumerate(alist) if k==key])), (lambda key, alist: # getalistの実体 (lambda tmp:tmp[0] if tmp != [] else None)( [e[1] for e in alist if key == e[0]])), [] # alistの実体 )
出力
1 2 fuga 2 [1, 2]
ほぼ手続き型ライクに使える。ただし、リストの要素などには直接代入できないので、そういったことに気を配る必要があるが(リストの要素に代入するだけならinsertしてpopすれば良いので関数で書けそう)。
もう少し頑張れば、可読性の高い(アンダーバー付きのメソッドを呼ばなくて良い)pythonワンライナーを書けそうだ。
【python】defaultdictは使い方をミスると重くて遅い
defaultdictはpythonで使えるとても便利なコレクション型です。
しかし、使い方には注意が必要な場合があります。
目次
defaultdictの概要
これはご存じない方向けの章なので、「知ってるよ」という方は読み飛ばしても構いません。
defaultdictは標準ライブラリのcollectionsモジュールに含まれるデータ型です。辞書に存在しないキーにアクセスしたとき、デフォルト値が設定されたキーを自動で作ってくれるのがメリットです。
次のように使えます。
>>> dict()["hoge"] # 通常のdictは存在しないキーにアクセスすると例外を吐く Traceback (most recent call last): File "<stdin>", line 1, in <module> KeyError: 'hoge' >>> from collections import defaultdict >>> d = defaultdict(int) >>> d["hoge"] # defaultdictはこうできる 0 >>> d["fuga"] += 1 # こんなことも >>> d["fuga"] 1
とても便利そうですが、アクセスするだけでキーが生成されてしまうということは問題を引き起こすことがあります。
問題点
こんなコードを実行してみます。
# coding: UTF-8 from collections import defaultdict def main(): d = defaultdict(str) d[0] = "hoge" d[10] = "fuga" d[100] = "piyo" lst = [d[i] for i in range(10000000)] print(len(d.keys())) if __name__ == "__main__": main()
タスクマネージャーなどでメモリ消費を監視しながら実行を見守ってください。けっこうなメモリが消費された後、10000000という数字が出てきます。
line_profilerで確認してみます(mainの前に@profileデコレータを付ける必要があります)。
$ kernprof -l -v test_defaultdict.py
10000000
Wrote profile results to test_defaultdict.py.lprof
Timer unit: 1e-06 s
Total time: 5.97851 s
File: test_defaultdict.py
Function: main at line 5
Line # Hits Time Per Hit % Time Line Contents
==============================================================
5 @profile
6 def main():
7 1 8.0 8.0 0.0 d = defaultdict(str)
8
9 1 2.0 2.0 0.0 d[0] = "hoge"
10 1 1.0 1.0 0.0 d[10] = "fuga"
11 1 1.0 1.0 0.0 d[100] = "piyo"
12
13 1 5978354.0 5978354.0 100.0 lst = [d[i] for i in range(10000000)]
14 1 141.0 141.0 0.0 print(len(d.keys()))やはり恐ろしいほど時間がかかっています。
この処理時間の大半は、キーの生成に費やされているはずです。メモリ消費が膨れ上がるのも、default値を持つキーを大量に作ったからdictオブジェクトが膨れ上がった、と説明できます。
つまり、こういう処理を書くとリーズナブルではないと言えます。
解決策
defaultdictと言えども、単に初期値を期待して参照するようなときはキーのあるなしを判定するべきです。
# coding: UTF-8 from collections import defaultdict def main(): d = defaultdict(str) d[0] = "hoge" d[10] = "fuga" d[100] = "piyo" lst = [d[i] if i in d else "" for i in range(10000000)] print(len(d.keys())) if __name__ == "__main__": main()
今度は心なしか少ない実行時間で3と表示され、メモリ消費も膨れ上がらないはずです。line_profilerで見ても、
$ kernprof -l -v test_defaultdict.py
3
Wrote profile results to test_defaultdict.py.lprof
Timer unit: 1e-06 s
Total time: 2.5628 s
File: test_defaultdict.py
Function: main at line 5
Line # Hits Time Per Hit % Time Line Contents
==============================================================
5 @profile
6 def main():
7 1 5.0 5.0 0.0 d = defaultdict(str)
8
9 1 1.0 1.0 0.0 d[0] = "hoge"
10 1 1.0 1.0 0.0 d[10] = "fuga"
11 1 1.0 1.0 0.0 d[100] = "piyo"
12
13 1 2562684.0 2562684.0 100.0 lst = [d[i] if i in d else "" for i in range(10000000)]
14 1 108.0 108.0 0.0 print(len(d.keys()))このように処理時間が半分以下になりました。
まとめ
- defaultdictは単にデフォルト値を期待してアクセスしただけの場合でも、hashにキーと値を追加してしまう。
- 通常のdictと同様、キーの存在を判定して処理を分岐させた方がリーズナブルである。
【python】pythonで主成分分析のバイプロット
バイプロット(Biplot)という主成分分析(PCA)の結果の可視化方法があります。
すごく大雑把に言うと、PCAによる写像の前の空間の各特徴(軸)が写像先の空間のどこに向いているかを可視化する方法です。
具体的には、主成分ベクトル(因子負荷量などを使う場合もあります)と散布図を同じ図にplotします。これらを組み合わせることで、元の空間の性質が二次元(もしかしたら3次元)で手に取るようにわかります*1。
バイプロットはR言語だと簡単に描けるらしいのですが、我らがpythonには(少なくとも一般的なライブラリには)そんな便利なものはありません。ちょっと困るのですが、幸い英語圏にはちらほらやりかたの情報があります。しかし、それはそれでページごとにやってることが違ったりして、(申し訳ないのですが)微妙に信用できなかったりします。
で、けっきょく自分で書いてみることにしました。なお、参考にしたのはこの辺です。
- GitHub - teddyroland/python-biplot: Generates simple biplot using common scientific Python packages
- Biplot with Python | SukhbinderSingh.com
- http://okomestudio.net/biboroku/?p=2292
方針
まずsklearnの公式ドキュメントをできるだけ良く読み込みます。
sklearn.decomposition.PCA — scikit-learn 0.22.1 documentation
PCA.components_が固有ベクトルであり、データをセンタリングしてこれと掛けるとPCAの出力が出てくることは前回の記事で確認しました。
固有ベクトル行列が主成分*元のデータの特徴という形になっているとして、横に見ると負荷量(みたいなもの。本当は対応する固有値のsqrtを掛け算してやらないといけない)に、縦に見ると元の写像先で表現された特徴の軸になります。
つまり、その軸をプロットするだけです。
なお、この辺は微妙に議論があるようです。私もこれがどこまで正しい方法なのかは自信が持てません。
参考:
色々と考えてみる: 文系のための「主成分分析の可視化」(2)
だけど今回は、データをセンタリングしてPCAを学習させた上で、各軸に対応するone-hot vectorを渡してtransformしたら確かに上に書いた方法通りで上手く行きました(biplotの線の上に載った)。なので、「これで良いんだろう」と勝手に判断しました。どこまで妥当かはよくわからないんですけど。
実装
こんな感じで書きました。
# coding: UTF-8 from sklearn.datasets import load_iris, load_wine from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA import matplotlib.pyplot as plt def biplot(dataset, scale=False, arrow_mul=1, text_mul=1.1): if scale: ss = StandardScaler() X = ss.fit_transform(dataset.data) else: X = dataset.data if hasattr(dataset, "feature_names"): feature_names = list(dataset.feature_names) else: feature_names = ["F{0}".format(i) for i in range(dataset.data.shape[1])] pca = PCA(n_components=2) X = pca.fit_transform(X) x_data = X[:,0] y_data = X[:,1] pc0 = pca.components_[0] pc1 = pca.components_[1] plt.figure() plt.scatter(x_data, y_data, c=dataset.target/len(set(dataset.target)), marker=".") for i in range(pc0.shape[0]): plt.arrow(0, 0, pc0[i]*arrow_mul, pc1[i]*arrow_mul, color='r') plt.text(pc0[i]*arrow_mul*text_mul, pc1[i]*arrow_mul*text_mul, feature_names[i], color='r') plt.show() def main(): iris = load_iris() wine = load_wine() biplot(iris, arrow_mul=2.5, scale=True) biplot(wine, arrow_mul=6, scale=True) if __name__ == "__main__": main()
今回はsklearnのデータセットを渡す形で関数にまとめました。ま、もしこのコードを流用したい人がいたら、必要なロジックだけ上手く切り出してください。
結果は、こんな画像が出ます。

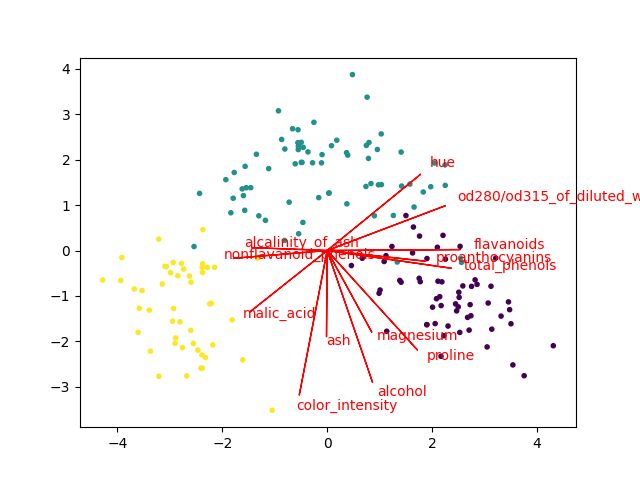
上手く行ってる感じです。
なお、上のコードでは変数をスケーリングしています(相関行列でPCAするのと等価)。スケーリングしなくてもできますが、やった方が矢印の長さが揃いやすいです(逆に変数のスケールを重視してPCAしたいときは、スケーリングしてはいけない。ケースバイケース)。
まとめ
これくらい自作しなくても済めば良いのにと思いました。
*1:本当に手に取るようにわかるかはデータと見る人に依存しますが・・・
【python】numpyで主成分分析を実装してみた
numpyでPCA(principal component analysis:主成分分析)を実装してみました。自分の理解を深めるためです。
sklearnに実装されているものと同じ結果を出すことを目標にしました。最終的には上手く行きました。
目次
概要
主成分分析のアルゴリズムの解説は他に譲ります。これは実装してみた記事です。
実装のやり方は色々あるようですが、一番基本的な(だと思う)共分散行列の固有値と固有ベクトルを求める方法で行きます。
やるべきこととしては、
- データをセンタリングする(列ごとに平均を引く)
- 共分散行列を計算する
- 固有値と固有ベクトルを計算
- データを固有ベクトルを使って写像する
これらを実装すれば行けるはずです。というか、これで行くことにしました。
実装
書いたソースコードを以下に示します。
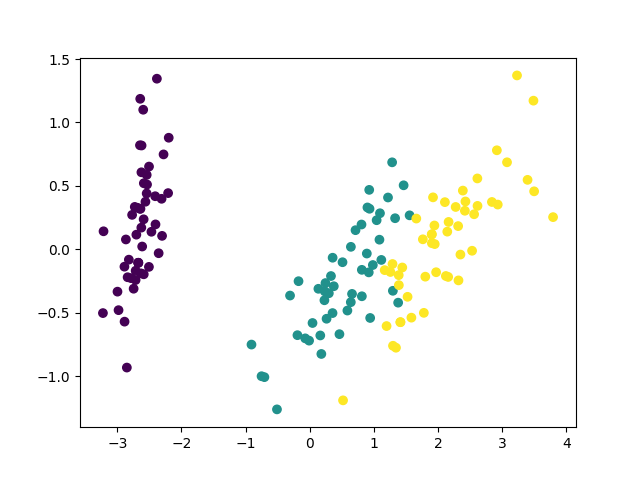
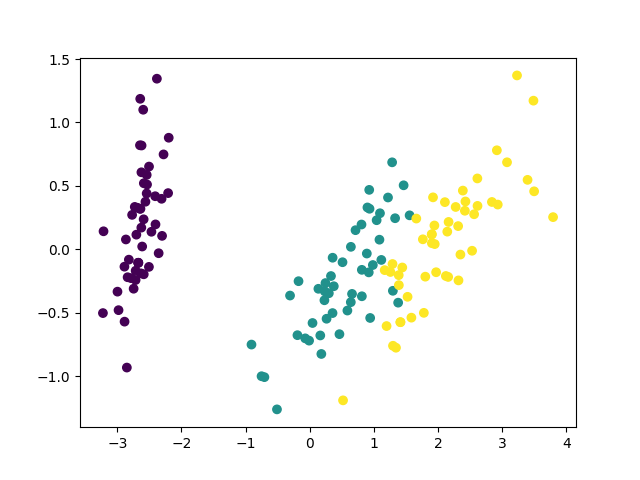
# coding: UTF-8 import numpy as np from sklearn.datasets import load_iris from sklearn.decomposition import PCA import matplotlib.pyplot as plt class MyPCA: def __init__(self, n_components=2): self.n_components = n_components def fit_transform(self, X): """横着してfit_transformしか実装してない """ # 平均を0にする X = X - X.mean(axis=0) # 共分散行列を作る self.cov_ = np.cov(X, rowvar=False) # 固有値と固有ベクトルを求めて固有値の大きい順にソート l, v = np.linalg.eig(self.cov_) l_index = np.argsort(l)[::-1] self.l_ = l[l_index] self.v_ = v[:,l_index] # 列ベクトルなのに注意 # components_(固有ベクトル行列を途中まで取り出す)を作る self.components_ = self.v_[:,:self.n_components].T # データとcomponents_をかける # 上と下で二回転置してるのアホ・・・ T = (np.mat(X)*(np.mat(self.components_.T))).A # 出力 return T def main(): iris = load_iris() pca = PCA(n_components=2) sklearn_X = pca.fit_transform(iris.data) my_pca = MyPCA() my_X = my_pca.fit_transform(iris.data) print(pca.explained_variance_) print(my_pca.l_) print(pca.components_) print(my_pca.components_) plt.figure() plt.scatter(sklearn_X[:,0], sklearn_X[:,1], c=iris.target/3) plt.savefig("sklearn_resut.png") plt.figure() plt.scatter(my_X[:,0], my_X[:,1]*-1, c=iris.target/3) plt.savefig("my_result.png") if __name__ == "__main__": main()
numpyを使ったので簡単に書けました。アルゴリズム部分はコメントで解説を入れたので、それを読めばどんな感じかは理解して頂けると思います。
結果
mainのテキスト出力を見ると、次のようになっていました。
# 固有値 [4.22484077 0.24224357] [4.22484077 0.24224357 0.07852391 0.02368303] # components_ [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [ 0.65653988 0.72971237 -0.1757674 -0.07470647]] [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [-0.65653988 -0.72971237 0.1757674 0.07470647]]
固有値が余計に出ちゃってますが、これは別に構いません。また、componentsの2次元目が符号反転していますが、これも特に問題ないこと(のはず)なので無視します。
自作の方は第二主成分を反転させてプロットしてみました。
同じ図を2つ載せるなって怒られそうですが・・・とにかく上手く行ったようです。
まとめ
numpyで実装してみたら思ったより簡単だったので、これで当分は「わかった気」になれそうです。
ただ、今回は特異値分解やらなかったので、それはまた宿題ということで・・・。
【python】カーネル主成分分析を試してみる
カーネル主成分分析(Kernel PCA)はカーネル法と主成分分析を組み合わせて用い、データを非線形次元圧縮する方法です(こんな説明で良いのか・・・)。
カーネル法のことは勉強中・・・というか正直勉強しようとしてもよくわからないで跳ね返されるのをこれまで4回くらい繰り返してきたのですが、とりあえず使ってみました。
試してみた
非線形データが手元にあると良いのですが、あいにくありません。輪っか状のデータなどを生成してやってみるのは簡単にできますが、面白くなさそうです。だいたいsklearnの公式サンプルにすらあります。
Kernel PCA — scikit-learn 0.21.2 documentation
そこで、分類問題での適用を考えます。これならいつものようにPCA+CLFとKPCA+CLFで比較するだけなので、簡単そうです。更に、カーネルのgammaはグリッドサーチして最適値を探すだけ・・・。
ただし、irisやdigitsで散々色々試してみましたが、ぶっちゃけ普通にやるとなかなかPCAを上回る性能が得られませんでした。最終的に、「digitsを3次元に次元削減し、LDAで分類する」という問題でどうにかそれなりに性能が上回ることがわかりましたが、実用的な意味はあまりありません。
たぶん、sklearnのtoy datasetは低次元で線形分離できるタチの良いデータばっかりなのだと思います。それはそれで良いことですが、ちょっとタチの悪いデータも混ぜておいてもらえると嬉しいところです(かといって20newsgroupsのBoWだとタチが悪すぎるし・・・2000データ400次元くらいのちょうど良いデータはどこかにないものか)。
コードを以下に示します。
# coding: UTF-8 import numpy as np from sklearn.datasets import load_digits from sklearn.decomposition import PCA, KernelPCA as KPCA from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA from sklearn.pipeline import Pipeline from sklearn.model_selection import GridSearchCV, StratifiedKFold as SKF from sklearn.model_selection import cross_val_score def main(): dataset = load_digits() print(dataset.data.shape) pca = PCA(n_components=3) kpca = KPCA(kernel="rbf", n_components=3) lda = LDA() pl_pca = Pipeline([("pca", pca), ("lda", lda)]) pl_kpca = Pipeline([("kpca", kpca), ("lda", lda)]) parameters = {"kpca__gamma" : np.arange(0.00001, 0.003, 0.0001)} clf = GridSearchCV(pl_kpca, parameters, verbose=0, n_jobs=-1) print(cross_val_score(pl_pca, dataset.data, dataset.target, cv=SKF(shuffle=True, random_state=0), scoring="f1_macro").mean()) print(cross_val_score(clf, dataset.data, dataset.target, cv=SKF(shuffle=True, random_state=0), scoring="f1_macro").mean()) if __name__ == "__main__": main()
PCAでは0.68らい、KPCAでは0.71くらいのF1値が得られました。
だから? って言われると、返す言葉は思いつきませんが・・・。
まとめ
やってみた記事ですが、何かの参考になればと思います。意外と上手く使うのは難しいと感じました。というか分類の次元削減としてはたぶんそんなに適当ではないです。
どんな問題に応用されてるんだろうか。やっぱり可視化?
追記
文字列の編集距離の可視化に使ってみました。
文字列カーネルというのもあるらしいのですが、sklearnで対応していないし、未確認。編集距離を使う分には無難に使えます。
【python】SelectKBestのscore_funcによる速度差を比較
SelectKBestはsklearnの簡単に特徴選択ができるクラスです。ざっくりと特徴選択したいときに、とても便利です。
sklearn.feature_selection.SelectKBest — scikit-learn 0.20.1 documentation
ところで、このSelectKBestにはscore_funcというパラメータを指定できます。このscore_funcには以下の選択肢があります。
- f_classif
- mutual_info_classif
- chi2
- f_regression
- mutual_info_regression
つまり、これらのスコア関数で特徴の「良し悪し」をスコア化し、良い特徴を優先的に選択するのがSelectKBestの動作です。
率直な疑問は、「それぞれの速度と性能はどうなのか」ということです。ということで、計測しました。
目次
各score_funcの概要
ドキュメントに書いてあることをざっくり訳しました。
- f_classif
ANOVAのF値
- mutual_info_classif
「discrete target」に対する相互情報量
- chi2
カイ二乗
- f_regression
特徴とラベルの単回帰のF値
- mutual_info_regression
「continuous target」に対する相互情報量
私がなんとなくでも理解できるのは、この中の半分くらいです。しかし本筋から逸れるので、それぞれの内容については今後勉強していくこととし、とりあえず先に進みます。
計測
お断り
mutual_info_classifおよびmutual_info_regressionは、他の100倍くらい遅かったので検討対象から外しました。処理コストを下げるために特徴選択するのに、特徴選択で処理コスト食ってたら意味がありません。
よって、比較したのは残り3つです。
- f_classif
- chi2
- f_regression
計測条件
対象にしたデータは、20newsgroupsをsklearnのCountVectorizerでBoW表現に変換したものです。
そこから更にデータと特徴をランダムサンプリングし、異なる条件下での性能を比較しています。
ランダムサンプリングの条件は、データ数を1000, 4000, 8000の3段階、データの次元数を1500, 2000, 3000の3段階、kを500の1段階でそれぞれ変化させた9つの組み合わせとしました。kによる速度差はなかったので(すべての特徴に対してスコアを出すので当たり前か)、kは変化させていません。
計測したのは、以下の2つの数値です。
- 全対象データを用いてfit_transformしたときの所要時間
- SelectKBestとRandomForestClassifierをPipelineで繋ぎ、交差検証で求めたF1値
Pipelineはleakage対策に必須です。
fit_transformの所要時間はマシンによって変化します。ただし、見たところ並列化して高速に計算しているような様子はどのスコア関数でもなかったので、他のマシンでも結果がひっくり返るような大きな違いは出てこない可能性が大きいです。
ソースコード
計測に使ったソースコードを以下に示します。
# coding: UTF-8 import time from itertools import product import numpy as np import pandas as pd from sklearn.datasets import fetch_20newsgroups from sklearn.feature_extraction.text import CountVectorizer from sklearn.feature_selection import SelectKBest as SKB,\ f_classif, chi2, f_regression from sklearn.ensemble import RandomForestClassifier as RFC from sklearn.model_selection import StratifiedKFold as SKF from sklearn.pipeline import Pipeline from sklearn.metrics import precision_recall_fscore_support as prf def main(): news20 = fetch_20newsgroups() cv = CountVectorizer(min_df=0.003, max_df=0.5) matrix = cv.fit_transform(news20.data).toarray() rfc = RFC(n_estimators=30, n_jobs=-1) df = pd.DataFrame([], columns=["data_n", "data_dim", "k", "FC-time", "CHI2-time", "FR-time", "FC-F1", "CHI2-F1", "FR-F1"]) for data_n, data_dim, k in product( [1000, 4000, 8000], [1500, 2000, 3000], [500]): print(data_n, data_dim, k) # データをランダムサンプリング original_index = np.arange(matrix.shape[0]) np.random.shuffle(original_index) sample_index = original_index[:data_n] X, y = matrix[sample_index], news20.target[sample_index] # 次元をランダムサンプリング original_dim_index = np.arange(matrix.shape[1]) np.random.shuffle(original_dim_index) sample_dim_index = original_dim_index[:data_dim] X = X[:,sample_dim_index] time_list = [] f1_list = [] for func_name, func in zip(["FC", "CHI2", "FR"], [f_classif, chi2, f_regression]): skb = SKB(score_func=func, k=k) t1 = time.time() skb.fit_transform(X, y) t2 = time.time() time_list.append(t2-t1) print("{0}:{1:.5f}".format(func_name, t2-t1)) clf = Pipeline([("skb", skb), ("rfc", rfc)]) trues = [] preds = [] for train_index, test_index in SKF().split(X, y): clf.fit(X[train_index], y[train_index]) trues.append(y[test_index]) preds.append(clf.predict(X[test_index])) score = prf(np.hstack(trues), np.hstack(preds), average="macro") print(score) f1_list.append(score[2]) s = pd.Series([data_n, data_dim, k, *time_list, *f1_list], index=df.columns) df = df.append(s, ignore_index=True) print(df) with open("result.csv", "w") as f: f.write(df.to_csv(index=False)) if __name__ == "__main__": main()
結果
結果の表を示します。これはCSV出力した計測結果をexcelで加工したものです。
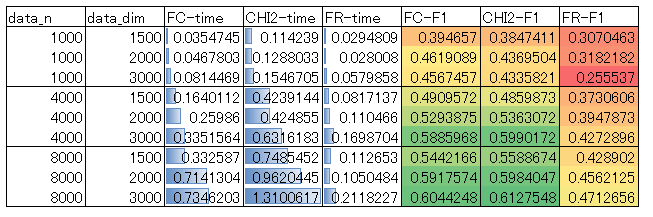
傾向を簡単に言うと、
処理時間は、f_regression<f_classif<chi2
性能は、f_regression<f_classif<=chi2
という結果になりました。
f_regressionは最速ですが、性能は悪いです。f_classifはf_regressionの数倍の時間がかかっていますが、性能はけっこう向上します。また、f_classifとchi2は性能の優劣がデータサイズなどで逆転するようですが、その差は微々たるものであり、chi2はf_classifの2~3倍の処理コストを要します。
結論
f_classifで良さそうです。デフォルトでそうなっているので、敢えて他のものを積極的に選ぶ理由は(特別な事情がない限り)なさそうです。
まとめ
f_classif(デフォルト)で良さそうでした。
【python】sklearnのPCAで相関行列を使う
主成分分析には共分散行列を用いる方法、相関行列を使う方法がある。
sklearnのPCAを見ると、これに対応するオプションは存在しない。
sklearn.decomposition.PCA — scikit-learn 0.20.1 documentation
ずっと不思議に思っていたが、ググってたらこんなものを見つけた。
要約:特徴量をスケーリングしてPCAすれば相関行列でやったのと同じことになるよ。PipelineでStandardScalerと組み合わせてね。おわり。
本当か確認する
確認してみる。
>>> import numpy as np >>> from sklearn.datasets import load_iris >>> from sklearn.preprocessing import StandardScaler >>> from sklearn.decomposition import PCA >>> from sklearn.pipeline import Pipeline >>> iris = load_iris() >>> pca = PCA(n_components=2) >>> pca.fit(iris.data) PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False) >>> pca.get_covariance() array([[ 0.67919741, -0.03258618, 1.27066452, 0.5321852 ], [-0.03258618, 0.18113034, -0.31863564, -0.13363564], [ 1.27066452, -0.31863564, 3.11934547, 1.28541527], [ 0.5321852 , -0.13363564, 1.28541527, 0.58961806]]) >>> ss = StandardScaler() >>> p = Pipeline([("scaler", ss), ("pca", pca)]) >>> p.fit(iris.data) Pipeline(memory=None, steps=[('scaler', StandardScaler(copy=True, with_mean=True, with_std=True)), ('pca', PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False))]) >>> p.steps[1][1].get_covariance() array([[ 0.9779242 , -0.10104477, 0.87069468, 0.86134879], [-0.10104477, 1.00395722, -0.41916911, -0.37286994], [ 0.87069468, -0.41916911, 1.04639367, 0.93676197], [ 0.86134879, -0.37286994, 0.93676197, 0.99857055]]) >>> np.corrcoef(iris.data, rowvar=False) array([[ 1. , -0.10936925, 0.87175416, 0.81795363], [-0.10936925, 1. , -0.4205161 , -0.35654409], [ 0.87175416, -0.4205161 , 1. , 0.9627571 ], [ 0.81795363, -0.35654409, 0.9627571 , 1. ]])
違うじゃん。妥当そうなのはnumpyの結果だが(対角成分が1になってる)、とりあえずしょうがないのでスケーリングしたデータの共分散をnumpyで計算してみる。
>>> np.cov(ss.fit_transform(iris.data), rowvar=0, bias=1) array([[ 1.00671141, -0.11010327, 0.87760486, 0.82344326], [-0.11010327, 1.00671141, -0.42333835, -0.358937 ], [ 0.87760486, -0.42333835, 1.00671141, 0.96921855], [ 0.82344326, -0.358937 , 0.96921855, 1.00671141]]) >>> np.cov(ss.fit_transform(iris.data), rowvar=0, bias=1) array([[ 1. , -0.10936925, 0.87175416, 0.81795363], [-0.10936925, 1. , -0.4205161 , -0.35654409], [ 0.87175416, -0.4205161 , 1. , 0.9627571 ], [ 0.81795363, -0.35654409, 0.9627571 , 1. ]])
標本分散はnp.corrcoefと等価だ。
ここまでやったところでもう一回ドキュメントを読み、PCA.get_covariance()の結果が「Estimated covariance of data.」であり、厳密ではないことに気づいたので、問題は解決した。
理論的にこうなる理由は、説明しようと思えばできるのだと思いますが、今回は大変なので触れません。
irisでやってみる
irisの可視化にそれぞれを使ってみる。コードを以下に示す。
# coding: UTF-8 from sklearn.datasets import load_iris from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA from sklearn.pipeline import Pipeline import matplotlib.pyplot as plt def main(): iris = load_iris() ss = StandardScaler() pca = PCA(n_components=2) p = Pipeline([("scaler", ss), ("pca", pca)]) X = pca.fit_transform(iris.data) plt.figure() plt.scatter(X[:,0], X[:,1], c=iris.target/3) plt.savefig("iris_cov_pca.png") X = p.fit_transform(iris.data) plt.figure() plt.scatter(X[:,0], X[:,1], c=iris.target/3) plt.savefig("iris_corr_pca.png") if __name__ == "__main__": main()
結果は、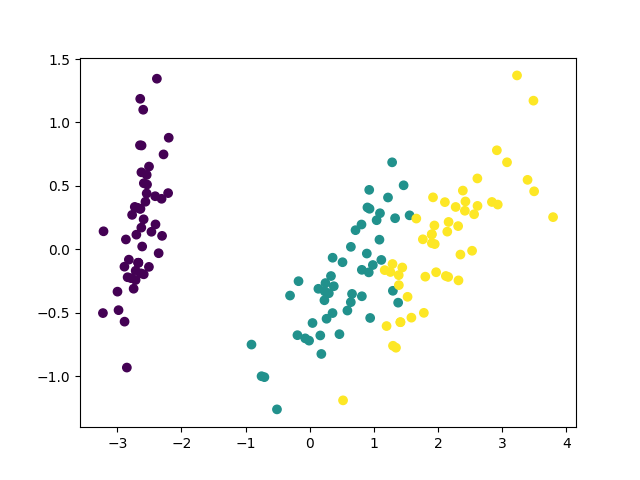
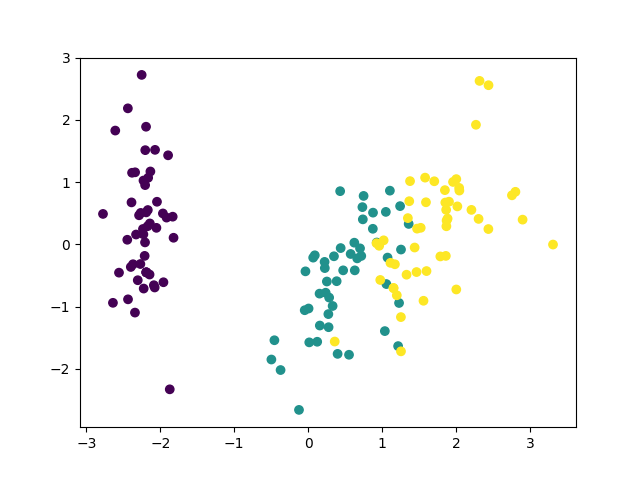
こうして見ると相関行列はあまりメリットがないように見えますが、実際には相関行列の方が良いタスクは色々あるようです。相関行列を使うことでbiplotが上手く行っているという例を出しているページを載せておきます。
PCA on correlation or covariance? - Cross Validated
まとめ
とりあえずできることはわかったので良しとする。
でも、「pipelineで出来るから要らねーよ」ってつもりらしいけど、ぶっちゃけオプション一つでできた方が親切だと思った(小並感)。
【python】sklearnのfetch_20newsgroupsで文書分類を試す(4)
前回は性能を追い求めると次元がでかくなりすぎて・・・というところで終わっていた。今回はもうちょっと頑張って次元を減らしてみる。
目次
ストップワードの除去
とりあえずstop_wordsを指定していなかったので、指定してみる。
stop_words="english"とすると、ストップワードを除去してくれる。
結果だけ言うと、min_df=0.005のとき、
- stop_words指定なし:3949次元
- stop_words指定あり:3705次元
だった。焼石に水。
PCA(主成分分析)とLDA(線形判別分析)
PCAとLDAをかけ、次元削減をする。leakage怖いのでPipelineを使う(厳密なことを言い出すと、単語文書行列を作る段からPipelineに入れるべきなのだろうか? きついのでパスさせて頂くが)。
PCAは主にLDAの計算負荷削減と、変数の相関を除去することを意図してかける。1000次元まで落としてみたが、これでも累積寄与率は90%弱になる。まあ、正規化も何もしてないから、重要な情報を落としている可能性は否定できないのだが。
LDAは次元削減に使う。有効性についてはこの前試してみたので、この記事を読んで欲しい。
【python】LDA(線形判別分析)で次元削減 - 静かなる名辞
20newsgroupsは20クラスのデータなので、19次元に落とすことになる。相当早くなるだろうが、どこまで性能を維持できるかはデータの線形性にかかっている。
分類
ランダムフォレストを使った。n_estimators=1000とし、他のパラメタはデフォルト。
ソースコード
実験に使ったソースコードを以下に示す。
# coding: UTF-8 import numpy as np from sklearn.datasets import fetch_20newsgroups from sklearn.ensemble import RandomForestClassifier as RFC from sklearn.feature_extraction.text import CountVectorizer as CV from sklearn.decomposition import PCA from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA from sklearn.model_selection import StratifiedKFold from sklearn.metrics import precision_recall_fscore_support as prf from sklearn.pipeline import Pipeline def main(): news20 = fetch_20newsgroups() cv = CV(min_df=0.005, max_df=0.5, stop_words="english") matrix = cv.fit_transform(news20.data).toarray() pca = PCA(n_components=1000, svd_solver="randomized") lda = LDA() rfc = RFC(n_estimators=1000, n_jobs=-1) clf = Pipeline([("pca", pca), ("lda", lda), ("rfc", rfc)]) trues = [] preds = [] for train_index, test_index in StratifiedKFold().split(matrix, news20.target): clf.fit(matrix[train_index], news20.target[train_index]) trues.append(news20.target[test_index]) preds.append(clf.predict(matrix[test_index])) scores = prf(np.hstack(trues), np.hstack(preds), average="macro")[:3] print("p:{0:.6f} r:{1:.6f} f1:{2:.6f}".format(scores[0], scores[1], scores[2])) if __name__ == "__main__": main()
結果とまとめ
p:0.764012 r:0.760731 f1:0.761510
前回の0.8を超えるスコアには届かなかったが、とりあえずそれなりに軽くはなった。もうちょっと真面目に追い込めばという話はあるが、追求しない。次回はもうちょっと違うことをやってみたい。
次回
このシリーズずっと放置していましたが、気が向いたので書きました。
www.haya-programming.com
