【python】numpyで主成分分析を実装してみた
numpyでPCA(principal component analysis:主成分分析)を実装してみました。自分の理解を深めるためです。
sklearnに実装されているものと同じ結果を出すことを目標にしました。最終的には上手く行きました。
目次
概要
主成分分析のアルゴリズムの解説は他に譲ります。これは実装してみた記事です。
実装のやり方は色々あるようですが、一番基本的な(だと思う)共分散行列の固有値と固有ベクトルを求める方法で行きます。
やるべきこととしては、
- データをセンタリングする(列ごとに平均を引く)
- 共分散行列を計算する
- 固有値と固有ベクトルを計算
- データを固有ベクトルを使って写像する
これらを実装すれば行けるはずです。というか、これで行くことにしました。
実装
書いたソースコードを以下に示します。
# coding: UTF-8 import numpy as np from sklearn.datasets import load_iris from sklearn.decomposition import PCA import matplotlib.pyplot as plt class MyPCA: def __init__(self, n_components=2): self.n_components = n_components def fit_transform(self, X): """横着してfit_transformしか実装してない """ # 平均を0にする X = X - X.mean(axis=0) # 共分散行列を作る self.cov_ = np.cov(X, rowvar=False) # 固有値と固有ベクトルを求めて固有値の大きい順にソート l, v = np.linalg.eig(self.cov_) l_index = np.argsort(l)[::-1] self.l_ = l[l_index] self.v_ = v[:,l_index] # 列ベクトルなのに注意 # components_(固有ベクトル行列を途中まで取り出す)を作る self.components_ = self.v_[:,:self.n_components].T # データとcomponents_をかける # 上と下で二回転置してるのアホ・・・ T = (np.mat(X)*(np.mat(self.components_.T))).A # 出力 return T def main(): iris = load_iris() pca = PCA(n_components=2) sklearn_X = pca.fit_transform(iris.data) my_pca = MyPCA() my_X = my_pca.fit_transform(iris.data) print(pca.explained_variance_) print(my_pca.l_) print(pca.components_) print(my_pca.components_) plt.figure() plt.scatter(sklearn_X[:,0], sklearn_X[:,1], c=iris.target/3) plt.savefig("sklearn_resut.png") plt.figure() plt.scatter(my_X[:,0], my_X[:,1]*-1, c=iris.target/3) plt.savefig("my_result.png") if __name__ == "__main__": main()
numpyを使ったので簡単に書けました。アルゴリズム部分はコメントで解説を入れたので、それを読めばどんな感じかは理解して頂けると思います。
結果
mainのテキスト出力を見ると、次のようになっていました。
# 固有値 [4.22484077 0.24224357] [4.22484077 0.24224357 0.07852391 0.02368303] # components_ [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [ 0.65653988 0.72971237 -0.1757674 -0.07470647]] [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [-0.65653988 -0.72971237 0.1757674 0.07470647]]
固有値が余計に出ちゃってますが、これは別に構いません。また、componentsの2次元目が符号反転していますが、これも特に問題ないこと(のはず)なので無視します。
自作の方は第二主成分を反転させてプロットしてみました。
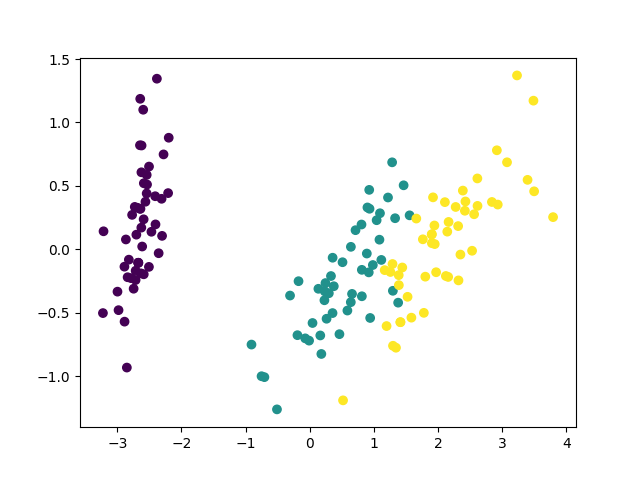
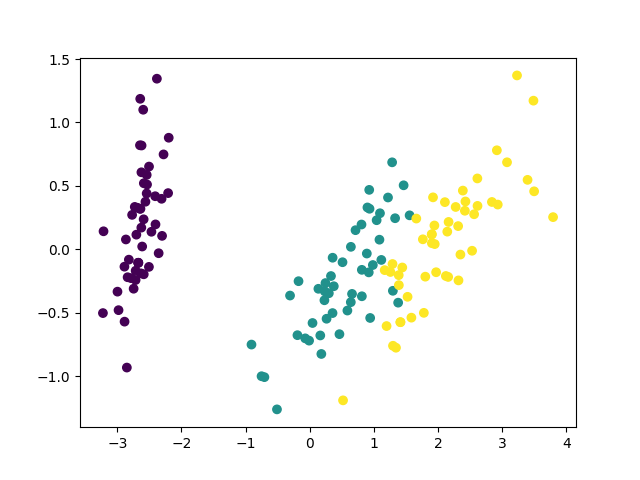
同じ図を2つ載せるなって怒られそうですが・・・とにかく上手く行ったようです。
まとめ
numpyで実装してみたら思ったより簡単だったので、これで当分は「わかった気」になれそうです。
ただ、今回は特異値分解やらなかったので、それはまた宿題ということで・・・。