【python】pythonで主成分分析のバイプロット
バイプロット(Biplot)という主成分分析(PCA)の結果の可視化方法があります。
すごく大雑把に言うと、PCAによる写像の前の空間の各特徴(軸)が写像先の空間のどこに向いているかを可視化する方法です。
具体的には、主成分ベクトル(因子負荷量などを使う場合もあります)と散布図を同じ図にplotします。これらを組み合わせることで、元の空間の性質が二次元(もしかしたら3次元)で手に取るようにわかります*1。
バイプロットはR言語だと簡単に描けるらしいのですが、我らがpythonには(少なくとも一般的なライブラリには)そんな便利なものはありません。ちょっと困るのですが、幸い英語圏にはちらほらやりかたの情報があります。しかし、それはそれでページごとにやってることが違ったりして、(申し訳ないのですが)微妙に信用できなかったりします。
で、けっきょく自分で書いてみることにしました。なお、参考にしたのはこの辺です。
- GitHub - teddyroland/python-biplot: Generates simple biplot using common scientific Python packages
- Biplot with Python | SukhbinderSingh.com
- http://okomestudio.net/biboroku/?p=2292
方針
まずsklearnの公式ドキュメントをできるだけ良く読み込みます。
sklearn.decomposition.PCA — scikit-learn 0.22.1 documentation
PCA.components_が固有ベクトルであり、データをセンタリングしてこれと掛けるとPCAの出力が出てくることは前回の記事で確認しました。
固有ベクトル行列が主成分*元のデータの特徴という形になっているとして、横に見ると負荷量(みたいなもの。本当は対応する固有値のsqrtを掛け算してやらないといけない)に、縦に見ると元の写像先で表現された特徴の軸になります。
つまり、その軸をプロットするだけです。
なお、この辺は微妙に議論があるようです。私もこれがどこまで正しい方法なのかは自信が持てません。
参考:
色々と考えてみる: 文系のための「主成分分析の可視化」(2)
だけど今回は、データをセンタリングしてPCAを学習させた上で、各軸に対応するone-hot vectorを渡してtransformしたら確かに上に書いた方法通りで上手く行きました(biplotの線の上に載った)。なので、「これで良いんだろう」と勝手に判断しました。どこまで妥当かはよくわからないんですけど。
実装
こんな感じで書きました。
# coding: UTF-8 from sklearn.datasets import load_iris, load_wine from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA import matplotlib.pyplot as plt def biplot(dataset, scale=False, arrow_mul=1, text_mul=1.1): if scale: ss = StandardScaler() X = ss.fit_transform(dataset.data) else: X = dataset.data if hasattr(dataset, "feature_names"): feature_names = list(dataset.feature_names) else: feature_names = ["F{0}".format(i) for i in range(dataset.data.shape[1])] pca = PCA(n_components=2) X = pca.fit_transform(X) x_data = X[:,0] y_data = X[:,1] pc0 = pca.components_[0] pc1 = pca.components_[1] plt.figure() plt.scatter(x_data, y_data, c=dataset.target/len(set(dataset.target)), marker=".") for i in range(pc0.shape[0]): plt.arrow(0, 0, pc0[i]*arrow_mul, pc1[i]*arrow_mul, color='r') plt.text(pc0[i]*arrow_mul*text_mul, pc1[i]*arrow_mul*text_mul, feature_names[i], color='r') plt.show() def main(): iris = load_iris() wine = load_wine() biplot(iris, arrow_mul=2.5, scale=True) biplot(wine, arrow_mul=6, scale=True) if __name__ == "__main__": main()
今回はsklearnのデータセットを渡す形で関数にまとめました。ま、もしこのコードを流用したい人がいたら、必要なロジックだけ上手く切り出してください。
結果は、こんな画像が出ます。

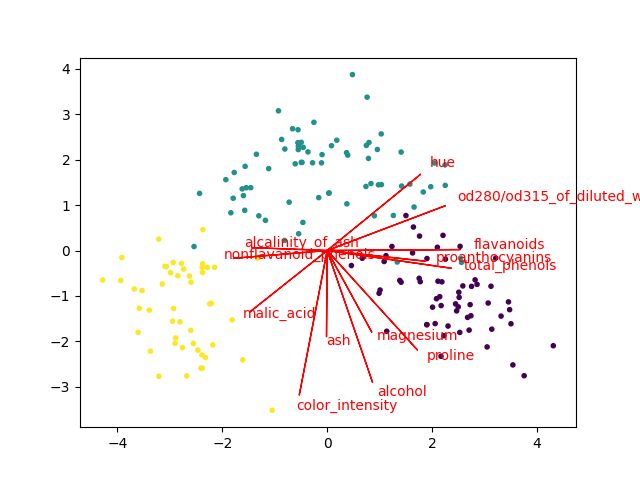
上手く行ってる感じです。
なお、上のコードでは変数をスケーリングしています(相関行列でPCAするのと等価)。スケーリングしなくてもできますが、やった方が矢印の長さが揃いやすいです(逆に変数のスケールを重視してPCAしたいときは、スケーリングしてはいけない。ケースバイケース)。
まとめ
これくらい自作しなくても済めば良いのにと思いました。
*1:本当に手に取るようにわかるかはデータと見る人に依存しますが・・・