【python】SelectKBestのscore_funcによる速度差を比較
SelectKBestはsklearnの簡単に特徴選択ができるクラスです。ざっくりと特徴選択したいときに、とても便利です。
sklearn.feature_selection.SelectKBest — scikit-learn 0.20.1 documentation
ところで、このSelectKBestにはscore_funcというパラメータを指定できます。このscore_funcには以下の選択肢があります。
- f_classif
- mutual_info_classif
- chi2
- f_regression
- mutual_info_regression
つまり、これらのスコア関数で特徴の「良し悪し」をスコア化し、良い特徴を優先的に選択するのがSelectKBestの動作です。
率直な疑問は、「それぞれの速度と性能はどうなのか」ということです。ということで、計測しました。
目次
各score_funcの概要
ドキュメントに書いてあることをざっくり訳しました。
- f_classif
ANOVAのF値
- mutual_info_classif
「discrete target」に対する相互情報量
- chi2
カイ二乗
- f_regression
特徴とラベルの単回帰のF値
- mutual_info_regression
「continuous target」に対する相互情報量
私がなんとなくでも理解できるのは、この中の半分くらいです。しかし本筋から逸れるので、それぞれの内容については今後勉強していくこととし、とりあえず先に進みます。
計測
お断り
mutual_info_classifおよびmutual_info_regressionは、他の100倍くらい遅かったので検討対象から外しました。処理コストを下げるために特徴選択するのに、特徴選択で処理コスト食ってたら意味がありません。
よって、比較したのは残り3つです。
- f_classif
- chi2
- f_regression
計測条件
対象にしたデータは、20newsgroupsをsklearnのCountVectorizerでBoW表現に変換したものです。
そこから更にデータと特徴をランダムサンプリングし、異なる条件下での性能を比較しています。
ランダムサンプリングの条件は、データ数を1000, 4000, 8000の3段階、データの次元数を1500, 2000, 3000の3段階、kを500の1段階でそれぞれ変化させた9つの組み合わせとしました。kによる速度差はなかったので(すべての特徴に対してスコアを出すので当たり前か)、kは変化させていません。
計測したのは、以下の2つの数値です。
- 全対象データを用いてfit_transformしたときの所要時間
- SelectKBestとRandomForestClassifierをPipelineで繋ぎ、交差検証で求めたF1値
Pipelineはleakage対策に必須です。
fit_transformの所要時間はマシンによって変化します。ただし、見たところ並列化して高速に計算しているような様子はどのスコア関数でもなかったので、他のマシンでも結果がひっくり返るような大きな違いは出てこない可能性が大きいです。
ソースコード
計測に使ったソースコードを以下に示します。
# coding: UTF-8 import time from itertools import product import numpy as np import pandas as pd from sklearn.datasets import fetch_20newsgroups from sklearn.feature_extraction.text import CountVectorizer from sklearn.feature_selection import SelectKBest as SKB,\ f_classif, chi2, f_regression from sklearn.ensemble import RandomForestClassifier as RFC from sklearn.model_selection import StratifiedKFold as SKF from sklearn.pipeline import Pipeline from sklearn.metrics import precision_recall_fscore_support as prf def main(): news20 = fetch_20newsgroups() cv = CountVectorizer(min_df=0.003, max_df=0.5) matrix = cv.fit_transform(news20.data).toarray() rfc = RFC(n_estimators=30, n_jobs=-1) df = pd.DataFrame([], columns=["data_n", "data_dim", "k", "FC-time", "CHI2-time", "FR-time", "FC-F1", "CHI2-F1", "FR-F1"]) for data_n, data_dim, k in product( [1000, 4000, 8000], [1500, 2000, 3000], [500]): print(data_n, data_dim, k) # データをランダムサンプリング original_index = np.arange(matrix.shape[0]) np.random.shuffle(original_index) sample_index = original_index[:data_n] X, y = matrix[sample_index], news20.target[sample_index] # 次元をランダムサンプリング original_dim_index = np.arange(matrix.shape[1]) np.random.shuffle(original_dim_index) sample_dim_index = original_dim_index[:data_dim] X = X[:,sample_dim_index] time_list = [] f1_list = [] for func_name, func in zip(["FC", "CHI2", "FR"], [f_classif, chi2, f_regression]): skb = SKB(score_func=func, k=k) t1 = time.time() skb.fit_transform(X, y) t2 = time.time() time_list.append(t2-t1) print("{0}:{1:.5f}".format(func_name, t2-t1)) clf = Pipeline([("skb", skb), ("rfc", rfc)]) trues = [] preds = [] for train_index, test_index in SKF().split(X, y): clf.fit(X[train_index], y[train_index]) trues.append(y[test_index]) preds.append(clf.predict(X[test_index])) score = prf(np.hstack(trues), np.hstack(preds), average="macro") print(score) f1_list.append(score[2]) s = pd.Series([data_n, data_dim, k, *time_list, *f1_list], index=df.columns) df = df.append(s, ignore_index=True) print(df) with open("result.csv", "w") as f: f.write(df.to_csv(index=False)) if __name__ == "__main__": main()
結果
結果の表を示します。これはCSV出力した計測結果をexcelで加工したものです。
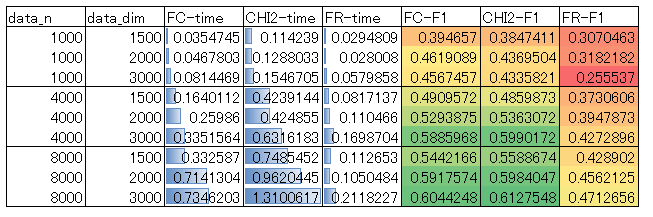
傾向を簡単に言うと、
処理時間は、f_regression<f_classif<chi2
性能は、f_regression<f_classif<=chi2
という結果になりました。
f_regressionは最速ですが、性能は悪いです。f_classifはf_regressionの数倍の時間がかかっていますが、性能はけっこう向上します。また、f_classifとchi2は性能の優劣がデータサイズなどで逆転するようですが、その差は微々たるものであり、chi2はf_classifの2~3倍の処理コストを要します。
結論
f_classifで良さそうです。デフォルトでそうなっているので、敢えて他のものを積極的に選ぶ理由は(特別な事情がない限り)なさそうです。
まとめ
f_classif(デフォルト)で良さそうでした。