【python】defaultdictは使い方をミスると重くて遅い
defaultdictはpythonで使えるとても便利なコレクション型です。
しかし、使い方には注意が必要な場合があります。
目次
defaultdictの概要
これはご存じない方向けの章なので、「知ってるよ」という方は読み飛ばしても構いません。
defaultdictは標準ライブラリのcollectionsモジュールに含まれるデータ型です。辞書に存在しないキーにアクセスしたとき、デフォルト値が設定されたキーを自動で作ってくれるのがメリットです。
次のように使えます。
>>> dict()["hoge"] # 通常のdictは存在しないキーにアクセスすると例外を吐く Traceback (most recent call last): File "<stdin>", line 1, in <module> KeyError: 'hoge' >>> from collections import defaultdict >>> d = defaultdict(int) >>> d["hoge"] # defaultdictはこうできる 0 >>> d["fuga"] += 1 # こんなことも >>> d["fuga"] 1
とても便利そうですが、アクセスするだけでキーが生成されてしまうということは問題を引き起こすことがあります。
問題点
こんなコードを実行してみます。
# coding: UTF-8 from collections import defaultdict def main(): d = defaultdict(str) d[0] = "hoge" d[10] = "fuga" d[100] = "piyo" lst = [d[i] for i in range(10000000)] print(len(d.keys())) if __name__ == "__main__": main()
タスクマネージャーなどでメモリ消費を監視しながら実行を見守ってください。けっこうなメモリが消費された後、10000000という数字が出てきます。
line_profilerで確認してみます(mainの前に@profileデコレータを付ける必要があります)。
$ kernprof -l -v test_defaultdict.py
10000000
Wrote profile results to test_defaultdict.py.lprof
Timer unit: 1e-06 s
Total time: 5.97851 s
File: test_defaultdict.py
Function: main at line 5
Line # Hits Time Per Hit % Time Line Contents
==============================================================
5 @profile
6 def main():
7 1 8.0 8.0 0.0 d = defaultdict(str)
8
9 1 2.0 2.0 0.0 d[0] = "hoge"
10 1 1.0 1.0 0.0 d[10] = "fuga"
11 1 1.0 1.0 0.0 d[100] = "piyo"
12
13 1 5978354.0 5978354.0 100.0 lst = [d[i] for i in range(10000000)]
14 1 141.0 141.0 0.0 print(len(d.keys()))やはり恐ろしいほど時間がかかっています。
この処理時間の大半は、キーの生成に費やされているはずです。メモリ消費が膨れ上がるのも、default値を持つキーを大量に作ったからdictオブジェクトが膨れ上がった、と説明できます。
つまり、こういう処理を書くとリーズナブルではないと言えます。
解決策
defaultdictと言えども、単に初期値を期待して参照するようなときはキーのあるなしを判定するべきです。
# coding: UTF-8 from collections import defaultdict def main(): d = defaultdict(str) d[0] = "hoge" d[10] = "fuga" d[100] = "piyo" lst = [d[i] if i in d else "" for i in range(10000000)] print(len(d.keys())) if __name__ == "__main__": main()
今度は心なしか少ない実行時間で3と表示され、メモリ消費も膨れ上がらないはずです。line_profilerで見ても、
$ kernprof -l -v test_defaultdict.py
3
Wrote profile results to test_defaultdict.py.lprof
Timer unit: 1e-06 s
Total time: 2.5628 s
File: test_defaultdict.py
Function: main at line 5
Line # Hits Time Per Hit % Time Line Contents
==============================================================
5 @profile
6 def main():
7 1 5.0 5.0 0.0 d = defaultdict(str)
8
9 1 1.0 1.0 0.0 d[0] = "hoge"
10 1 1.0 1.0 0.0 d[10] = "fuga"
11 1 1.0 1.0 0.0 d[100] = "piyo"
12
13 1 2562684.0 2562684.0 100.0 lst = [d[i] if i in d else "" for i in range(10000000)]
14 1 108.0 108.0 0.0 print(len(d.keys()))このように処理時間が半分以下になりました。
まとめ
- defaultdictは単にデフォルト値を期待してアクセスしただけの場合でも、hashにキーと値を追加してしまう。
- 通常のdictと同様、キーの存在を判定して処理を分岐させた方がリーズナブルである。
【python】pythonで主成分分析のバイプロット
バイプロット(Biplot)という主成分分析(PCA)の結果の可視化方法があります。
すごく大雑把に言うと、PCAによる写像の前の空間の各特徴(軸)が写像先の空間のどこに向いているかを可視化する方法です。
具体的には、主成分ベクトル(因子負荷量などを使う場合もあります)と散布図を同じ図にplotします。これらを組み合わせることで、元の空間の性質が二次元(もしかしたら3次元)で手に取るようにわかります*1。
バイプロットはR言語だと簡単に描けるらしいのですが、我らがpythonには(少なくとも一般的なライブラリには)そんな便利なものはありません。ちょっと困るのですが、幸い英語圏にはちらほらやりかたの情報があります。しかし、それはそれでページごとにやってることが違ったりして、(申し訳ないのですが)微妙に信用できなかったりします。
で、けっきょく自分で書いてみることにしました。なお、参考にしたのはこの辺です。
- GitHub - teddyroland/python-biplot: Generates simple biplot using common scientific Python packages
- Biplot with Python | SukhbinderSingh.com
- http://okomestudio.net/biboroku/?p=2292
方針
まずsklearnの公式ドキュメントをできるだけ良く読み込みます。
sklearn.decomposition.PCA — scikit-learn 0.22.1 documentation
PCA.components_が固有ベクトルであり、データをセンタリングしてこれと掛けるとPCAの出力が出てくることは前回の記事で確認しました。
固有ベクトル行列が主成分*元のデータの特徴という形になっているとして、横に見ると負荷量(みたいなもの。本当は対応する固有値のsqrtを掛け算してやらないといけない)に、縦に見ると元の写像先で表現された特徴の軸になります。
つまり、その軸をプロットするだけです。
なお、この辺は微妙に議論があるようです。私もこれがどこまで正しい方法なのかは自信が持てません。
参考:
色々と考えてみる: 文系のための「主成分分析の可視化」(2)
だけど今回は、データをセンタリングしてPCAを学習させた上で、各軸に対応するone-hot vectorを渡してtransformしたら確かに上に書いた方法通りで上手く行きました(biplotの線の上に載った)。なので、「これで良いんだろう」と勝手に判断しました。どこまで妥当かはよくわからないんですけど。
実装
こんな感じで書きました。
# coding: UTF-8 from sklearn.datasets import load_iris, load_wine from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA import matplotlib.pyplot as plt def biplot(dataset, scale=False, arrow_mul=1, text_mul=1.1): if scale: ss = StandardScaler() X = ss.fit_transform(dataset.data) else: X = dataset.data if hasattr(dataset, "feature_names"): feature_names = list(dataset.feature_names) else: feature_names = ["F{0}".format(i) for i in range(dataset.data.shape[1])] pca = PCA(n_components=2) X = pca.fit_transform(X) x_data = X[:,0] y_data = X[:,1] pc0 = pca.components_[0] pc1 = pca.components_[1] plt.figure() plt.scatter(x_data, y_data, c=dataset.target/len(set(dataset.target)), marker=".") for i in range(pc0.shape[0]): plt.arrow(0, 0, pc0[i]*arrow_mul, pc1[i]*arrow_mul, color='r') plt.text(pc0[i]*arrow_mul*text_mul, pc1[i]*arrow_mul*text_mul, feature_names[i], color='r') plt.show() def main(): iris = load_iris() wine = load_wine() biplot(iris, arrow_mul=2.5, scale=True) biplot(wine, arrow_mul=6, scale=True) if __name__ == "__main__": main()
今回はsklearnのデータセットを渡す形で関数にまとめました。ま、もしこのコードを流用したい人がいたら、必要なロジックだけ上手く切り出してください。
結果は、こんな画像が出ます。

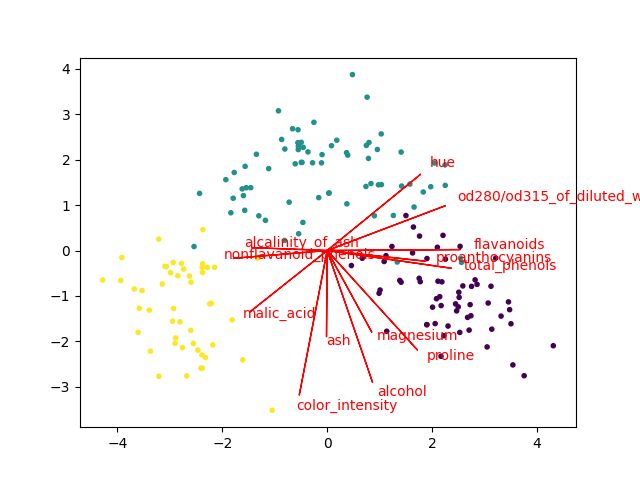
上手く行ってる感じです。
なお、上のコードでは変数をスケーリングしています(相関行列でPCAするのと等価)。スケーリングしなくてもできますが、やった方が矢印の長さが揃いやすいです(逆に変数のスケールを重視してPCAしたいときは、スケーリングしてはいけない。ケースバイケース)。
まとめ
これくらい自作しなくても済めば良いのにと思いました。
*1:本当に手に取るようにわかるかはデータと見る人に依存しますが・・・
【python】numpyで主成分分析を実装してみた
numpyでPCA(principal component analysis:主成分分析)を実装してみました。自分の理解を深めるためです。
sklearnに実装されているものと同じ結果を出すことを目標にしました。最終的には上手く行きました。
目次
概要
主成分分析のアルゴリズムの解説は他に譲ります。これは実装してみた記事です。
実装のやり方は色々あるようですが、一番基本的な(だと思う)共分散行列の固有値と固有ベクトルを求める方法で行きます。
やるべきこととしては、
- データをセンタリングする(列ごとに平均を引く)
- 共分散行列を計算する
- 固有値と固有ベクトルを計算
- データを固有ベクトルを使って写像する
これらを実装すれば行けるはずです。というか、これで行くことにしました。
実装
書いたソースコードを以下に示します。
# coding: UTF-8 import numpy as np from sklearn.datasets import load_iris from sklearn.decomposition import PCA import matplotlib.pyplot as plt class MyPCA: def __init__(self, n_components=2): self.n_components = n_components def fit_transform(self, X): """横着してfit_transformしか実装してない """ # 平均を0にする X = X - X.mean(axis=0) # 共分散行列を作る self.cov_ = np.cov(X, rowvar=False) # 固有値と固有ベクトルを求めて固有値の大きい順にソート l, v = np.linalg.eig(self.cov_) l_index = np.argsort(l)[::-1] self.l_ = l[l_index] self.v_ = v[:,l_index] # 列ベクトルなのに注意 # components_(固有ベクトル行列を途中まで取り出す)を作る self.components_ = self.v_[:,:self.n_components].T # データとcomponents_をかける # 上と下で二回転置してるのアホ・・・ T = (np.mat(X)*(np.mat(self.components_.T))).A # 出力 return T def main(): iris = load_iris() pca = PCA(n_components=2) sklearn_X = pca.fit_transform(iris.data) my_pca = MyPCA() my_X = my_pca.fit_transform(iris.data) print(pca.explained_variance_) print(my_pca.l_) print(pca.components_) print(my_pca.components_) plt.figure() plt.scatter(sklearn_X[:,0], sklearn_X[:,1], c=iris.target/3) plt.savefig("sklearn_resut.png") plt.figure() plt.scatter(my_X[:,0], my_X[:,1]*-1, c=iris.target/3) plt.savefig("my_result.png") if __name__ == "__main__": main()
numpyを使ったので簡単に書けました。アルゴリズム部分はコメントで解説を入れたので、それを読めばどんな感じかは理解して頂けると思います。
結果
mainのテキスト出力を見ると、次のようになっていました。
# 固有値 [4.22484077 0.24224357] [4.22484077 0.24224357 0.07852391 0.02368303] # components_ [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [ 0.65653988 0.72971237 -0.1757674 -0.07470647]] [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [-0.65653988 -0.72971237 0.1757674 0.07470647]]
固有値が余計に出ちゃってますが、これは別に構いません。また、componentsの2次元目が符号反転していますが、これも特に問題ないこと(のはず)なので無視します。
自作の方は第二主成分を反転させてプロットしてみました。
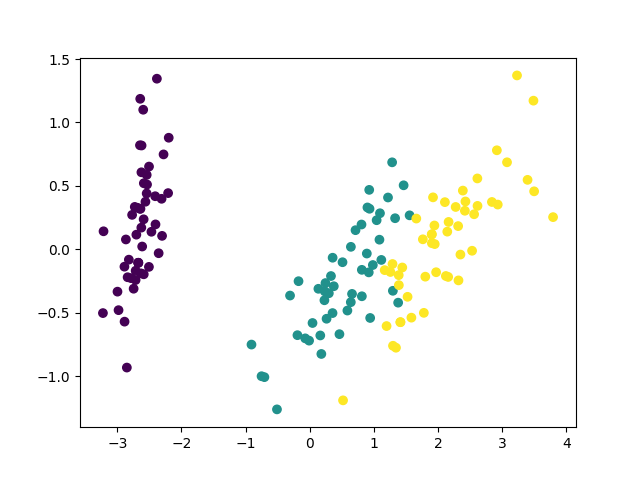
同じ図を2つ載せるなって怒られそうですが・・・とにかく上手く行ったようです。
まとめ
numpyで実装してみたら思ったより簡単だったので、これで当分は「わかった気」になれそうです。
ただ、今回は特異値分解やらなかったので、それはまた宿題ということで・・・。
【python】カーネル主成分分析を試してみる
カーネル主成分分析(Kernel PCA)はカーネル法と主成分分析を組み合わせて用い、データを非線形次元圧縮する方法です(こんな説明で良いのか・・・)。
カーネル法のことは勉強中・・・というか正直勉強しようとしてもよくわからないで跳ね返されるのをこれまで4回くらい繰り返してきたのですが、とりあえず使ってみました。
試してみた
非線形データが手元にあると良いのですが、あいにくありません。輪っか状のデータなどを生成してやってみるのは簡単にできますが、面白くなさそうです。だいたいsklearnの公式サンプルにすらあります。
Kernel PCA — scikit-learn 0.21.2 documentation
そこで、分類問題での適用を考えます。これならいつものようにPCA+CLFとKPCA+CLFで比較するだけなので、簡単そうです。更に、カーネルのgammaはグリッドサーチして最適値を探すだけ・・・。
ただし、irisやdigitsで散々色々試してみましたが、ぶっちゃけ普通にやるとなかなかPCAを上回る性能が得られませんでした。最終的に、「digitsを3次元に次元削減し、LDAで分類する」という問題でどうにかそれなりに性能が上回ることがわかりましたが、実用的な意味はあまりありません。
たぶん、sklearnのtoy datasetは低次元で線形分離できるタチの良いデータばっかりなのだと思います。それはそれで良いことですが、ちょっとタチの悪いデータも混ぜておいてもらえると嬉しいところです(かといって20newsgroupsのBoWだとタチが悪すぎるし・・・2000データ400次元くらいのちょうど良いデータはどこかにないものか)。
コードを以下に示します。
# coding: UTF-8 import numpy as np from sklearn.datasets import load_digits from sklearn.decomposition import PCA, KernelPCA as KPCA from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA from sklearn.pipeline import Pipeline from sklearn.model_selection import GridSearchCV, StratifiedKFold as SKF from sklearn.model_selection import cross_val_score def main(): dataset = load_digits() print(dataset.data.shape) pca = PCA(n_components=3) kpca = KPCA(kernel="rbf", n_components=3) lda = LDA() pl_pca = Pipeline([("pca", pca), ("lda", lda)]) pl_kpca = Pipeline([("kpca", kpca), ("lda", lda)]) parameters = {"kpca__gamma" : np.arange(0.00001, 0.003, 0.0001)} clf = GridSearchCV(pl_kpca, parameters, verbose=0, n_jobs=-1) print(cross_val_score(pl_pca, dataset.data, dataset.target, cv=SKF(shuffle=True, random_state=0), scoring="f1_macro").mean()) print(cross_val_score(clf, dataset.data, dataset.target, cv=SKF(shuffle=True, random_state=0), scoring="f1_macro").mean()) if __name__ == "__main__": main()
PCAでは0.68らい、KPCAでは0.71くらいのF1値が得られました。
だから? って言われると、返す言葉は思いつきませんが・・・。
まとめ
やってみた記事ですが、何かの参考になればと思います。意外と上手く使うのは難しいと感じました。というか分類の次元削減としてはたぶんそんなに適当ではないです。
どんな問題に応用されてるんだろうか。やっぱり可視化?
追記
文字列の編集距離の可視化に使ってみました。
文字列カーネルというのもあるらしいのですが、sklearnで対応していないし、未確認。編集距離を使う分には無難に使えます。
【python】SelectKBestのscore_funcによる速度差を比較
SelectKBestはsklearnの簡単に特徴選択ができるクラスです。ざっくりと特徴選択したいときに、とても便利です。
sklearn.feature_selection.SelectKBest — scikit-learn 0.20.1 documentation
ところで、このSelectKBestにはscore_funcというパラメータを指定できます。このscore_funcには以下の選択肢があります。
- f_classif
- mutual_info_classif
- chi2
- f_regression
- mutual_info_regression
つまり、これらのスコア関数で特徴の「良し悪し」をスコア化し、良い特徴を優先的に選択するのがSelectKBestの動作です。
率直な疑問は、「それぞれの速度と性能はどうなのか」ということです。ということで、計測しました。
目次
各score_funcの概要
ドキュメントに書いてあることをざっくり訳しました。
- f_classif
ANOVAのF値
- mutual_info_classif
「discrete target」に対する相互情報量
- chi2
カイ二乗
- f_regression
特徴とラベルの単回帰のF値
- mutual_info_regression
「continuous target」に対する相互情報量
私がなんとなくでも理解できるのは、この中の半分くらいです。しかし本筋から逸れるので、それぞれの内容については今後勉強していくこととし、とりあえず先に進みます。
計測
お断り
mutual_info_classifおよびmutual_info_regressionは、他の100倍くらい遅かったので検討対象から外しました。処理コストを下げるために特徴選択するのに、特徴選択で処理コスト食ってたら意味がありません。
よって、比較したのは残り3つです。
- f_classif
- chi2
- f_regression
計測条件
対象にしたデータは、20newsgroupsをsklearnのCountVectorizerでBoW表現に変換したものです。
そこから更にデータと特徴をランダムサンプリングし、異なる条件下での性能を比較しています。
ランダムサンプリングの条件は、データ数を1000, 4000, 8000の3段階、データの次元数を1500, 2000, 3000の3段階、kを500の1段階でそれぞれ変化させた9つの組み合わせとしました。kによる速度差はなかったので(すべての特徴に対してスコアを出すので当たり前か)、kは変化させていません。
計測したのは、以下の2つの数値です。
- 全対象データを用いてfit_transformしたときの所要時間
- SelectKBestとRandomForestClassifierをPipelineで繋ぎ、交差検証で求めたF1値
Pipelineはleakage対策に必須です。
fit_transformの所要時間はマシンによって変化します。ただし、見たところ並列化して高速に計算しているような様子はどのスコア関数でもなかったので、他のマシンでも結果がひっくり返るような大きな違いは出てこない可能性が大きいです。
ソースコード
計測に使ったソースコードを以下に示します。
# coding: UTF-8 import time from itertools import product import numpy as np import pandas as pd from sklearn.datasets import fetch_20newsgroups from sklearn.feature_extraction.text import CountVectorizer from sklearn.feature_selection import SelectKBest as SKB,\ f_classif, chi2, f_regression from sklearn.ensemble import RandomForestClassifier as RFC from sklearn.model_selection import StratifiedKFold as SKF from sklearn.pipeline import Pipeline from sklearn.metrics import precision_recall_fscore_support as prf def main(): news20 = fetch_20newsgroups() cv = CountVectorizer(min_df=0.003, max_df=0.5) matrix = cv.fit_transform(news20.data).toarray() rfc = RFC(n_estimators=30, n_jobs=-1) df = pd.DataFrame([], columns=["data_n", "data_dim", "k", "FC-time", "CHI2-time", "FR-time", "FC-F1", "CHI2-F1", "FR-F1"]) for data_n, data_dim, k in product( [1000, 4000, 8000], [1500, 2000, 3000], [500]): print(data_n, data_dim, k) # データをランダムサンプリング original_index = np.arange(matrix.shape[0]) np.random.shuffle(original_index) sample_index = original_index[:data_n] X, y = matrix[sample_index], news20.target[sample_index] # 次元をランダムサンプリング original_dim_index = np.arange(matrix.shape[1]) np.random.shuffle(original_dim_index) sample_dim_index = original_dim_index[:data_dim] X = X[:,sample_dim_index] time_list = [] f1_list = [] for func_name, func in zip(["FC", "CHI2", "FR"], [f_classif, chi2, f_regression]): skb = SKB(score_func=func, k=k) t1 = time.time() skb.fit_transform(X, y) t2 = time.time() time_list.append(t2-t1) print("{0}:{1:.5f}".format(func_name, t2-t1)) clf = Pipeline([("skb", skb), ("rfc", rfc)]) trues = [] preds = [] for train_index, test_index in SKF().split(X, y): clf.fit(X[train_index], y[train_index]) trues.append(y[test_index]) preds.append(clf.predict(X[test_index])) score = prf(np.hstack(trues), np.hstack(preds), average="macro") print(score) f1_list.append(score[2]) s = pd.Series([data_n, data_dim, k, *time_list, *f1_list], index=df.columns) df = df.append(s, ignore_index=True) print(df) with open("result.csv", "w") as f: f.write(df.to_csv(index=False)) if __name__ == "__main__": main()
結果
結果の表を示します。これはCSV出力した計測結果をexcelで加工したものです。
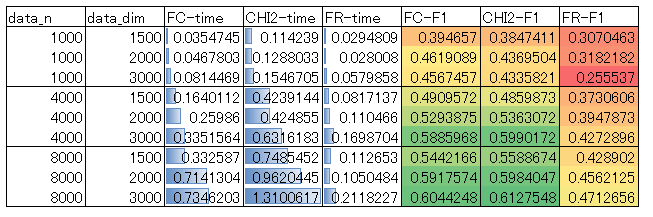
傾向を簡単に言うと、
処理時間は、f_regression<f_classif<chi2
性能は、f_regression<f_classif<=chi2
という結果になりました。
f_regressionは最速ですが、性能は悪いです。f_classifはf_regressionの数倍の時間がかかっていますが、性能はけっこう向上します。また、f_classifとchi2は性能の優劣がデータサイズなどで逆転するようですが、その差は微々たるものであり、chi2はf_classifの2~3倍の処理コストを要します。
結論
f_classifで良さそうです。デフォルトでそうなっているので、敢えて他のものを積極的に選ぶ理由は(特別な事情がない限り)なさそうです。
まとめ
f_classif(デフォルト)で良さそうでした。
【python】sklearnのPCAで相関行列を使う
主成分分析には共分散行列を用いる方法、相関行列を使う方法がある。
sklearnのPCAを見ると、これに対応するオプションは存在しない。
sklearn.decomposition.PCA — scikit-learn 0.20.1 documentation
ずっと不思議に思っていたが、ググってたらこんなものを見つけた。
要約:特徴量をスケーリングしてPCAすれば相関行列でやったのと同じことになるよ。PipelineでStandardScalerと組み合わせてね。おわり。
本当か確認する
確認してみる。
>>> import numpy as np >>> from sklearn.datasets import load_iris >>> from sklearn.preprocessing import StandardScaler >>> from sklearn.decomposition import PCA >>> from sklearn.pipeline import Pipeline >>> iris = load_iris() >>> pca = PCA(n_components=2) >>> pca.fit(iris.data) PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False) >>> pca.get_covariance() array([[ 0.67919741, -0.03258618, 1.27066452, 0.5321852 ], [-0.03258618, 0.18113034, -0.31863564, -0.13363564], [ 1.27066452, -0.31863564, 3.11934547, 1.28541527], [ 0.5321852 , -0.13363564, 1.28541527, 0.58961806]]) >>> ss = StandardScaler() >>> p = Pipeline([("scaler", ss), ("pca", pca)]) >>> p.fit(iris.data) Pipeline(memory=None, steps=[('scaler', StandardScaler(copy=True, with_mean=True, with_std=True)), ('pca', PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False))]) >>> p.steps[1][1].get_covariance() array([[ 0.9779242 , -0.10104477, 0.87069468, 0.86134879], [-0.10104477, 1.00395722, -0.41916911, -0.37286994], [ 0.87069468, -0.41916911, 1.04639367, 0.93676197], [ 0.86134879, -0.37286994, 0.93676197, 0.99857055]]) >>> np.corrcoef(iris.data, rowvar=False) array([[ 1. , -0.10936925, 0.87175416, 0.81795363], [-0.10936925, 1. , -0.4205161 , -0.35654409], [ 0.87175416, -0.4205161 , 1. , 0.9627571 ], [ 0.81795363, -0.35654409, 0.9627571 , 1. ]])
違うじゃん。妥当そうなのはnumpyの結果だが(対角成分が1になってる)、とりあえずしょうがないのでスケーリングしたデータの共分散をnumpyで計算してみる。
>>> np.cov(ss.fit_transform(iris.data), rowvar=0, bias=1) array([[ 1.00671141, -0.11010327, 0.87760486, 0.82344326], [-0.11010327, 1.00671141, -0.42333835, -0.358937 ], [ 0.87760486, -0.42333835, 1.00671141, 0.96921855], [ 0.82344326, -0.358937 , 0.96921855, 1.00671141]]) >>> np.cov(ss.fit_transform(iris.data), rowvar=0, bias=1) array([[ 1. , -0.10936925, 0.87175416, 0.81795363], [-0.10936925, 1. , -0.4205161 , -0.35654409], [ 0.87175416, -0.4205161 , 1. , 0.9627571 ], [ 0.81795363, -0.35654409, 0.9627571 , 1. ]])
標本分散はnp.corrcoefと等価だ。
ここまでやったところでもう一回ドキュメントを読み、PCA.get_covariance()の結果が「Estimated covariance of data.」であり、厳密ではないことに気づいたので、問題は解決した。
理論的にこうなる理由は、説明しようと思えばできるのだと思いますが、今回は大変なので触れません。
irisでやってみる
irisの可視化にそれぞれを使ってみる。コードを以下に示す。
# coding: UTF-8 from sklearn.datasets import load_iris from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA from sklearn.pipeline import Pipeline import matplotlib.pyplot as plt def main(): iris = load_iris() ss = StandardScaler() pca = PCA(n_components=2) p = Pipeline([("scaler", ss), ("pca", pca)]) X = pca.fit_transform(iris.data) plt.figure() plt.scatter(X[:,0], X[:,1], c=iris.target/3) plt.savefig("iris_cov_pca.png") X = p.fit_transform(iris.data) plt.figure() plt.scatter(X[:,0], X[:,1], c=iris.target/3) plt.savefig("iris_corr_pca.png") if __name__ == "__main__": main()
結果は、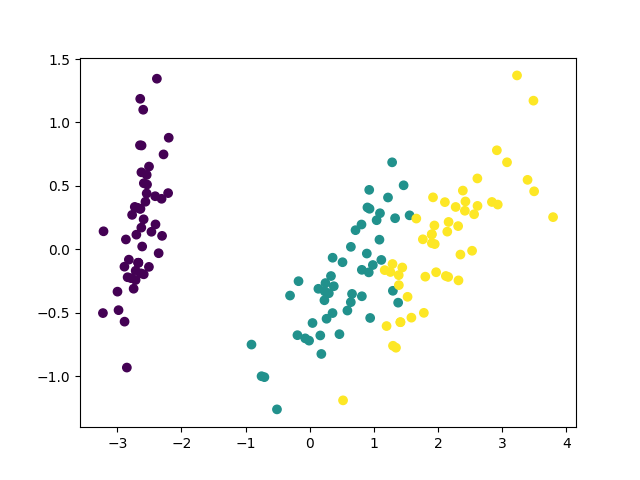
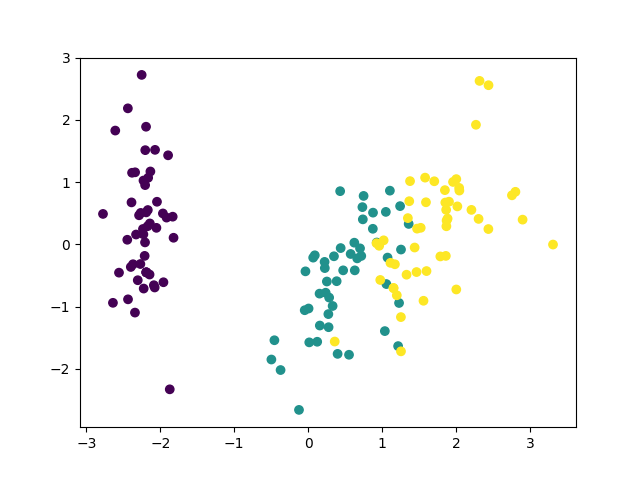
こうして見ると相関行列はあまりメリットがないように見えますが、実際には相関行列の方が良いタスクは色々あるようです。相関行列を使うことでbiplotが上手く行っているという例を出しているページを載せておきます。
PCA on correlation or covariance? - Cross Validated
まとめ
とりあえずできることはわかったので良しとする。
でも、「pipelineで出来るから要らねーよ」ってつもりらしいけど、ぶっちゃけオプション一つでできた方が親切だと思った(小並感)。
【python】sklearnのfetch_20newsgroupsで文書分類を試す(4)
前回は性能を追い求めると次元がでかくなりすぎて・・・というところで終わっていた。今回はもうちょっと頑張って次元を減らしてみる。
目次
ストップワードの除去
とりあえずstop_wordsを指定していなかったので、指定してみる。
stop_words="english"とすると、ストップワードを除去してくれる。
結果だけ言うと、min_df=0.005のとき、
- stop_words指定なし:3949次元
- stop_words指定あり:3705次元
だった。焼石に水。
PCA(主成分分析)とLDA(線形判別分析)
PCAとLDAをかけ、次元削減をする。leakage怖いのでPipelineを使う(厳密なことを言い出すと、単語文書行列を作る段からPipelineに入れるべきなのだろうか? きついのでパスさせて頂くが)。
PCAは主にLDAの計算負荷削減と、変数の相関を除去することを意図してかける。1000次元まで落としてみたが、これでも累積寄与率は90%弱になる。まあ、正規化も何もしてないから、重要な情報を落としている可能性は否定できないのだが。
LDAは次元削減に使う。有効性についてはこの前試してみたので、この記事を読んで欲しい。
【python】LDA(線形判別分析)で次元削減 - 静かなる名辞
20newsgroupsは20クラスのデータなので、19次元に落とすことになる。相当早くなるだろうが、どこまで性能を維持できるかはデータの線形性にかかっている。
分類
ランダムフォレストを使った。n_estimators=1000とし、他のパラメタはデフォルト。
ソースコード
実験に使ったソースコードを以下に示す。
# coding: UTF-8 import numpy as np from sklearn.datasets import fetch_20newsgroups from sklearn.ensemble import RandomForestClassifier as RFC from sklearn.feature_extraction.text import CountVectorizer as CV from sklearn.decomposition import PCA from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA from sklearn.model_selection import StratifiedKFold from sklearn.metrics import precision_recall_fscore_support as prf from sklearn.pipeline import Pipeline def main(): news20 = fetch_20newsgroups() cv = CV(min_df=0.005, max_df=0.5, stop_words="english") matrix = cv.fit_transform(news20.data).toarray() pca = PCA(n_components=1000, svd_solver="randomized") lda = LDA() rfc = RFC(n_estimators=1000, n_jobs=-1) clf = Pipeline([("pca", pca), ("lda", lda), ("rfc", rfc)]) trues = [] preds = [] for train_index, test_index in StratifiedKFold().split(matrix, news20.target): clf.fit(matrix[train_index], news20.target[train_index]) trues.append(news20.target[test_index]) preds.append(clf.predict(matrix[test_index])) scores = prf(np.hstack(trues), np.hstack(preds), average="macro")[:3] print("p:{0:.6f} r:{1:.6f} f1:{2:.6f}".format(scores[0], scores[1], scores[2])) if __name__ == "__main__": main()
結果とまとめ
p:0.764012 r:0.760731 f1:0.761510
前回の0.8を超えるスコアには届かなかったが、とりあえずそれなりに軽くはなった。もうちょっと真面目に追い込めばという話はあるが、追求しない。次回はもうちょっと違うことをやってみたい。
次回
このシリーズずっと放置していましたが、気が向いたので書きました。
www.haya-programming.com
【python】collections.ChainMapの使い方を理解する
はじめに
pythonで複数の辞書をマージするにはどうしたらいいのでしょうか。forループ? 辞書内包表記を使う? updateメソッド? 実は、ChainMapというものもあります。
その使い方について説明します。
先に結論
記事本文でだらだらと説明していますが、要約すると、
- 複数の辞書を一つにまとめる方法
- 元辞書への参照を張ってごにょごにょするだけなので、新しい辞書を作るより(ケースバイケースだが)速い。ただしマージが速くても参照が遅いという微妙な性質がある
- 当然元辞書を変更すると反映されてしまう
これだけです。
基本的な使い方
リストを渡すのかと思ったら、違うようです。
>>> from collections import ChainMap >>> d1 = {"hoge":"ほげ"} >>> d2 = {"fuga":"ふが"} >>> cm = ChainMap(d1, d2) >>> cm["hoge"] 'ほげ' >>> cm["fuga"] 'ふが'
このように使えます。ただ、辞書のリストを動的に生成して渡したい場合もあるでしょう。その場合、starred expressionを活用して、
>>> cm = ChainMap(*[d1, d2])
>>> cm
ChainMap({'hoge': 'ほげ'}, {'fuga': 'ふが'})
このようにやることになるかと思います。
参照先を変更してみる
>>> d1["HOGE"] = "ホゲ" >>> cm ChainMap({'hoge': 'ほげ', 'HOGE': 'ホゲ'}, {'fuga': 'ふが'})
このようにけっこう恐ろしい性質があります。これが嫌なときは他の方法で辞書をマージするか、deepcopyを使うことになるはずです。
速いらしいという噂があるので測る
測ってみました。上記参考サイトの辞書内包表記版と比較します。
予想では、マージは速いものの探索は恐らくhashのリストを線形探索していく上、重複を取り除く処理などもあるので、遅い要素がありそうです。
items()と一つのキーへのアクセスで近似的に探索を表現します。
# coding: UTF-8 import time from collections import ChainMap def time_measure(f, lst): time_lst = [] for i in range(10000): t1 = time.time() result = f(lst) t2 = time.time() time_lst.append(t2-t1) return result, sum(time_lst)/10000 def main(): f1 = lambda dicts:{k: v for dic in dicts for k, v in dic.items()} f2 = lambda dicts:ChainMap(*dicts) d1 = dict(zip(range(100), range(100))) d2 = dict(zip("ho", "ge")) d3 = dict(zip(range(100), "p"*100)) result, time = time_measure(f1, [d1, d2, d3]) print("辞書内包マージ", time) f = lambda _:[(k,v) for k,v in result.items()] _, time = time_measure(f, None) print("辞書内包items()", time) f = lambda _:result["h"] _, time = time_measure(f, None) print("辞書内包キーアクセス", time) result, time = time_measure(f2, [d1, d2, d3]) print("ChainMapマージ", time) f = lambda _:[(k,v) for k,v in result.items()] _, time = time_measure(f, None) print("ChainMap items()", time) f = lambda _:result["h"] _, time = time_measure(f, None) print("ChainMapキーアクセス", time) if __name__ == "__main__": main()
結果は
辞書内包マージ 1.2324166297912597e-05 辞書内包items() 7.125544548034668e-06 辞書内包キーアクセス 1.9309520721435546e-07 ChainMapマージ 1.3034582138061524e-06 ChainMap items() 6.01630687713623e-05 ChainMapキーアクセス 1.183009147644043e-06
マージは若干遅いが探索は速い辞書、マージは速いものの肝心の探索が遅いChainMapという結論。
探索速度(実タスクでの探索速度ではないが)に1桁の差が出てる訳で、安直に「ChainMap使おう」とはならないと思う・・・。普通はアクセスの速さを享受したいから辞書にすると思うのだが。
まとめ
使い所は難しいと思いました。
【python】外部プロセスと標準入出力で通信する
pythonで外部プロセス(subprocess)と、標準入出力を介したやりとりをしたいときがある。
目次
やってみること
今回は問題例として、形態素解析器MeCabをpythonから呼んでみる。MeCabはpythonバインディングがあるので、実用的には外部プロセスとして起動する必要はないのだが、わかりやすい例として敢えて挙げる。
なお、MeCabを知らない方のために説明すると、自然言語処理で使う形態素解析器というもので、次のような使い方をするものである。
$ mecab 吾輩は猫である。 # 入力行。この下の行から解析結果 吾輩 名詞,代名詞,一般,*,*,*,吾輩,ワガハイ,ワガハイ は 助詞,係助詞,*,*,*,*,は,ハ,ワ 猫 名詞,一般,*,*,*,*,猫,ネコ,ネコ で 助動詞,*,*,*,特殊・ダ,連用形,だ,デ,デ ある 助動詞,*,*,*,五段・ラ行アル,基本形,ある,アル,アル 。 記号,句点,*,*,*,*,。,。,。 EOS
pythonスクリプトから外部プロセスとしてMeCabを起動し、入力をpythonスクリプトから送り込み、解析結果を得ることを目標にする。
簡単な方法
とりあえずやる
標準ライブラリのsubprocessを用いる。次のように書く。
# coding: UTF-8 import subprocess def main(): input_string = "吾輩は猫である。" mecab_p = subprocess.Popen(["mecab"], stdin=subprocess.PIPE, stdout=subprocess.PIPE) output_string = mecab_p.communicate(input_string.encode())[0].decode() print(output_string) if __name__ == "__main__": main()
結果
吾輩 名詞,代名詞,一般,*,*,*,吾輩,ワガハイ,ワガハイ は 助詞,係助詞,*,*,*,*,は,ハ,ワ 猫 名詞,一般,*,*,*,*,猫,ネコ,ネコ で 助動詞,*,*,*,特殊・ダ,連用形,だ,デ,デ ある 助動詞,*,*,*,五段・ラ行アル,基本形,ある,アル,アル 。 記号,句点,*,*,*,*,。,。,。 EOS
できた。
解説
subprocess.Popenでプロセスを立ち上げパイプをつなぐ。後はmecab_p.communicateの引数で入力を、返り値で出力を得る。なお、返り値は
注意すべき点としては、端末から打ち込むのと基本的に同じなので文字コードを意識する必要がある。python内部で使われている文字列型は、(python3では)unicode文字列(str型)という「とりあえず文字コードを意識しなくても使える型」になっているのだが、外部プロセスに投げるときはencodeしてbytes型という要するにただのバイト列にしてやる必要があり、また外部プロセスから受け取った文字列もまたbytes型なのでdecodeしてstr型に戻してやらないといけない。
今回はlinux上の環境で実験しているので、文字コードはpython、システム、mecabすべてutf-8で統一されており、その場合は上のコードのように簡単に書ける。
windows環境だったりすると(けっきょくはpythonとmecabの設定に依存するが)、encodingが統一されていないこともあり得るので、そういうときはちゃんと自分で環境を設定した通りにencodingを取り扱ってやらないと、たぶん動かない。
応用:複数回入出力を送りつけたい
subprocess.Popen.communicateを2回呼ぶと、次のようなエラーが出る。
ValueError: Cannot send input after starting communication
つまり1回しかやり取りできない。送りつけて出力を受け取った瞬間、パイプは閉じられてしまう。安全といえば安全だが、これは不便である。
この回避のためには、
- 複数回の入力を一つにまとめて送りつける
- stdin, stdoutを直接叩く
といった方法がある、らしい。
参考:python - Communicate multiple times with a process without breaking the pipe? - Stack Overflow
前者の方法はそれで済めば良いだろうけど、たまには対応しきれないときもある。後者の方法は、そんな見るからにおっかない(制御ミスるとすぐ死ねそうな)方法は使いたくない。というか、実際やってみても上手く動かせなかった。
なので、Pexpectというライブラリに頼る。これは擬似端末を立ち上げてプロセスとおしゃべりする系のもの。コマンドの自動化などに使うものらしい。
結論から言うと、これを使うのはけっこう辛かった。挙動のクセが強すぎる。正規表現に慣れててexpectの世界観も理解してる人には良いんだろうけど、僕みたいな素人にはただただ望む出力を得るまで試行錯誤を重ねる苦行だった。
def main2(): inputs = ["吾輩は猫である。", "名前はまだない。"] p = pexpect.spawn("/bin/bash") #p.logfile_read = sys.stdout.buffer p.sendline("mecab") p.readline() p.sendline("") p.expect("EOS") results = [] for s in inputs: p.sendline(s) p.expect("EOS") results.append(p.before.decode()) p.terminate() p.expect(pexpect.EOF) for r in results: print(r)
うまい正規表現が思いつかなかったのでMeCabのEOSを取ることに。
なんだか微妙な結果になってしまったけど、Pexpect自体はたぶん良いライブラリなので、然るべき場面で活用してあげてください。標準入出力を自動化したいときはたぶん使えます。ただ、今回の用途にはいまいち合ってなかった気がする。
まとめ
パイプで投げつけるだけだろと思ってたら、けっこう色々なことに気を遣う羽目になりました。
【python】nltkで英語のStemmingとLemmatization
Stemming(ステミング)は単語の語幹を取り出したいとき、Lemmatization(レンマ化、敢えてカタカナ表記するとレンマタイゼーション)はカテゴリごとにグルーピングしたりしたいときに使う。
公式ドキュメントはここ。
nltk.stem package — NLTK 3.4 documentation
目次
スポンサーリンク
Stemming
概要
nltkでStemmingに使えるクラスはたくさんある(ように見える)。nltk.stemに実装されているものだけでも、
- ARLSTem Arabic Stemmer*1
アラビア語用
- ISRI Arabic Stemmer*2
アラビア語用
- Lancaster Stemmer*3
英語用。古い(1990年)
- Porter Stemmer*4
英語用。古い(1980年)
- Regexp Stemmer
正規表現で実装されている。どんな正規表現にするかは使うときに指定する
- RSLP Stemmer
ポルトガル語用
- Snowball Stemmers
なんか色々な言語に対応している
これだけある。注釈はぜんぶドキュメントからコピペした出典です。
とりあえず英語で何も考えずに使えるのは、Lancaster、Porter、Snowball Stemmersの3択。LancasterとPorterは基本的にルールベース、Snowball StemmersはPorter StemmerのPorterさんの作ったフレームワークで、多言語対応していることが特徴なのだけど肝心の英語版のアルゴリズムはPorterらしいので(ドキュメントにそう書いてある)、選択肢から外すと、けっきょくLancaster、Porterの2択になる。
なお、Stemmingの研究がどこまで進んでいるかはこのドキュメントを見て勉強した。
http://kenbenoit.net/assets/courses/tcd2014qta/readings/Jivani_ijcta2011020632.pdf
統計的言語処理に走って、けっきょくこれは難しいタスクというか、一単語ずつ見てもどうしようもないよね、という壁にぶち当たった・・・って感じか。今はどんな感じになってるんだろうか(というかstemming自体、今はどこまで重要視されているのだろうか)。一応、nltkに実装されているアルゴリズムも、ある程度の完成度には達しているのだろうけど。
nltk.stemで使えるアルゴリズムはそういった難しい問題が立ちはだかる以前のものなので、とりあえず気楽に使えると言えば使えそう。n-gramとかHMMとかを応用したStemmerも提案はされてるっぽいのだが、nltkで使えなさそうなので没。
Porterを使う
とりあえずPorterから先に使ってみる。
>>> from nltk.stem.porter import PorterStemmer as PS >>> ps = PS() >>> ps.stem("dogs") 'dog' >>> ps.stem("cats") 'cat' >>> ps.stem("ceiling") 'ceil'
これだけ。最後のceilingは「eg. ceil- is not the stem of ceiling」という例なので、解析がいまいち上手く行っていないということになる(この例はnltk.stemの公式ドキュメントの最後に出ている)。
Lancasterを使ってみる
>>> from nltk.stem.lancaster import LancasterStemmer as LS >>> ls = LS() >>> ls.stem("dogs") 'dog' >>> ls.stem("cats") 'cat' >>> ls.stem("ceiling") 'ceil'
まったく同様に使えるようだ。
Lemmatizing
nltk.stemのページの一番最後にしれっと載ってる。wordNetを使って単語をカテゴリに落としてくれる。一体どんな粒度でやってくれるのかよくわからないのだが、公式の例を見るとstemmingと同様に機能している気がする。
なお、初回呼び出し時は親切なことに、
LookupError:
**********************************************************************
Resource wordnet not found.
Please use the NLTK Downloader to obtain the resource:
>>> import nltk
>>> nltk.download('wordnet')こんなエラーを吐いてくれる。downloadすると使えるようになる。
>>> from nltk.stem.wordnet import WordNetLemmatizer as WNL >>> wnl = WNL() >>> wnl.lemmatize("dogs") 'dog' >>> wnl.lemmatize("cats") 'cat' >>> wnl.lemmatize("ceiling") 'ceiling'
Lemmatizer特有のメリットとしては、
>>> wnl.lemmatize("good", pos="a") 'good' >>> wnl.lemmatize("better", pos="a") 'good'
こういうことができる。この例はpos="a"(adjective)を指定しないと名詞として取り扱われて上手く行かないみたいだけど。このposキーワードについては、そもそもPOSタグ判定が成功しないと上手く使えない上、POS taggerの吐くPOSタグと指定できるPOSタグに互換性がなさそうなので、使い方はかなり難しそう。無理を重ねている感じ。
なお、公式曰く、「Returns the input word unchanged if it cannot be found in WordNet.」とのこと。つまり「元々語幹の形で入力されたのでchangeする必要がなかった」のか「見つからないからとりあえずそのまま返した」だけなのか区別がつかない。ふざけんなと言いたい。せめて検索して見つかったかどうかくらいはT/Fでも1/0でも良いから返してくれ。
ま、そういうことになっているので、実際どう使うべきなのかは率直に言って悩む。
結論
研究者・開発者の皆さんには申し訳ないけど、どれもどんぐりの背比べ感が否めない・・・。
*1:K. Abainia, S. Ouamour and H. Sayoud, A Novel Robust Arabic Light Stemmer , Journal of Experimental & Theoretical Artificial Intelligence (JETAI‘17), Vol. 29, No. 3, 2017, pp. 557-573.
*2:Taghva, K., Elkoury, R., and Coombs, J. 2005. Arabic Stemming without a root dictionary. Information Science Research Institute. University of Nevada, Las Vegas, USA.
*3:Paice, Chris D. “Another Stemmer.” ACM SIGIR Forum 24.3 (1990): 56-61.
*4:Porter, M. “An algorithm for suffix stripping.” Program 14.3 (1980): 130-137.
【python】matplotlibで3次元データを描画し、回転アニメーションにする
3次元くらいのデータを描画したいときがある。簡単に散布図にできると便利。
データの用意
sklearnのload_irisなどで取得できるデータセットを入力にする前提の次のような関数を作った。
from sklearn.decomposition import PCA def gen_3d_data(dataset): pca = PCA(n_components=3) return pca.fit_transform(dataset.data), dataset.target
あとはirisなり何なりを入れてやる。
スポンサーリンク
3次元プロット
とりあえずプロットしたいだけならこれだけ。
import matplotlib.pyplot as plt from mpl_toolkits.mplot3d import axes3d def matplotlib_plt(X, y, filename): fig = plt.figure() ax = fig.add_subplot(111, projection="3d") ax.scatter(X[:,0], X[:,1], X[:,2], c=y/len(set(y))) plt.savefig(filename) plt.show()
点の色の指定がちょっとセコいが、まあ良いこととする。axes3dはパっと見使ってないように見えるが、importしないとエラーになるので必要と思われる。
呼び出し元のmainも書く。
from sklearn.datasets import load_iris def main(): iris_X, iris_y = gen_3d_data(load_iris()) matplotlib_plt(iris_X, iris_y, "iris.png")
実行すると次のような画像が出力される。
回転させたアニメーションを表示
このような例が公式で示されている。
for angle in range(0, 360): ax.view_init(30, angle) plt.draw() plt.pause(.001)
mplot3d example code: rotate_axes3d_demo.py — Matplotlib 2.0.2 documentation
やると確かにぐるぐる回るアニメーションが表示される。こりゃあええわ、ということで、次はこれをgifアニメにすることを考える。
matplotlibにもanimationというモジュールがあり、色々できるようだが使い方を覚えるのが大変そうだった。なので、「上のforループ内で一枚ずつ画像出力してffmpegで繋げば良いだろ」という手抜きの方針で行くことにする。
def matplotlib_rotate(X, y, dataname): fig = plt.figure() ax = fig.add_subplot(111, projection="3d") ax.scatter(X[:,0], X[:,1], X[:,2], c=y/len(set(y))) for angle in range(0, 360): ax.view_init(30, angle) plt.savefig("figs/{0}_{1:03d}.jpg".format(dataname, angle))
呼び方は、
matplotlib_rotate(iris_X, iris_y, "iris")
こうするとfigs/以下に画像が360枚吐かれるので、ffmpegでつなぐ。
$ ffmpeg -r 10 -i figs/iris_%03d.jpg -pix_fmt rgb24 -f gif out.gif
とりあえずこれで行けた。画質が悪い割に容量が重いので、どこか上手くない指定になってるのかもしれないけど。

上出来ではないだろうか。
【python】flymakeのエラー
次のようなエラーを吐かれた。
Flymake: Configuration error has occurred while running(***/flymakes ***.py).Flymake will be switched OFF
原因
色々あるらしいけど、今回は文字コード指定をタイポした瞬間エラーが出た。
# coding: UTF-9
たぶん、これを書いた瞬間にflymakeが走って「そんな文字コードないよ!解釈できない!」→落ちる、という流れだったんだと思う。
対策
これも色んなサイトに書いてあることだが、次の設定をemacsの設定ファイルに付け足す。
(defadvice flymake-post-syntax-check (before flymake-force-check-was-interrupted) (setq flymake-check-was-interrupted t)) (ad-activate 'flymake-post-syntax-check)
参考にしたページ:
flymakeのsyntax-checkが異常終了しても無視するようにする - すぎゃーんメモ
とりあえずflymakeは落ちなくなったが、UTF-9とか書いちゃってもエラー表示してくれる訳ではないので、微妙っちゃ微妙。でもそれに対応させる気概もないのでこれで行く。
【python】関数オブジェクトは辞書のキーに使える!
「まさかできねーだろw」と思ってやったらできたのでびっくりしたよ!
>>> def hoge(): ... return None ... >>> {hoge:1} {<function hoge at 0x7f025f18cf28>: 1} ||< えぇ…。 lambdaでもできる。 >|python| >>> fuga = lambda:None >>> {fuga:2} {<function <lambda> at 0x7f0243880510>: 2}
hogeとfugaは関数として同値である(敢えてそう言えば)。ということは・・・!?
>>> hoge in {hoge:1} True >>> fuga in {hoge:1} False
よかった、ちょっとホッとした。
ん、待てよ・・・
>>> def piyo(): ... return None ... >>> piyo in {hoge:1} False
こっちもちゃんとFalseだった。よかった。
どうしてこうなるのか。
こちらの記事を読んだ。
Python における hashable - Qiita
__hash__メソッドがありさえすれば辞書のキーに使えるらしい。試してみよう。
>>> hoge.__hash__() -9223363308844643086 >>> fuga.__hash__() 8727981228113
あるんだ・・・。
>>> hoge.hogehoge = "hogee^~" >>> hoge.__hash__() -9223363308844643086
定義時に決まるimmutableだと思う。まあ、それは予想できてた。hashが更新できたら大騒ぎだ(関数の処理内容に応じて決まるのが本来のhashではないか? という気がしなくはないが。でも関数オブジェクトの処理内容は後から変更できる訳ではないので(高階関数で渡す等は除く)別に構わないのだろう)。
悪用方法
国際難読化pythonコードコンテストがあれば使えそう(ないけど)。たとえば、15秒くらいで思いついたちょっと格好良いコードはこんな感じ。
>>> d = None >>> d = {lambda :d:d}
lambdaのコロンとdictのコロンが混ざって非常に見た目が素敵。こんなのでもちゃんと構文解析されるので、pythonは偉い。
それ以外の使い道・・・keyを呼び出せるというのがすべてですが、それが活きそうな場面は思いつきそうで思いつかない。微妙な感じです。
30秒ほど悩んだところで「ヘタに使い道があると使うバカが出てきて危険」ということに思い至り、それ以上考えることをやめました。
【python】cabochaのpythonバインディングの変な挙動
環境
ubuntu 14.04
cabocha 0.69
cabocha-python 0.69
問題の概要
変な挙動だった。というか率直に言ってバグなのでは?
>>> import CaboCha >>> cparser = CaboCha.Parser() >>> tree1 = cparser.parse("吾輩は猫である。") >>> print(tree1.toString(CaboCha.FORMAT_TREE)) 吾輩は-D 猫である。 EOS >>> tree2 = cparser.parse("吾輩は猫ではない。") >>> print(tree1.toString(CaboCha.FORMAT_TREE)) 吾輩は-D 猫ではない。 EOS
これはおかしい。CaboChaはこわれている。
いや、「これで仕様通り動いてる。おまえの使い方が間違ってるんだ」て言われたら反論できないけど。詳細なドキュメントを見かけたことがないので、もしかしたらアホなこと(Parserのインスタンスの使い回し)をやっているのかもしれない。
回避するために試したこと
- CaboCha.Parser("")する(コンストラクタに空文字列を渡す)
効果なし。
- 一度空文字列に対してparseを呼ぶ
ここを参考に「もしかしたら効くかも」と思ってやってみた。
MeCabのparseToNodeのひどいバグ - 北野坂備忘録
効果なし。
- 文字列を変数に入れる、encodeする
ここを参考に(以下略)。
MeCabをPythonから使う注意点とか
効果なし。encodeに至ってはやったら落ちた。
- 仕方がないのでCaboCha.Parser()を毎回作る
>>> tree = CaboCha.Parser().parse("吾輩は猫である。")
>>> tree.toString(CaboCha.FORMAT_TREE)
Segmentation fault (コアダンプ)
たぶん本体のメモリ管理とpythonの接合が上手く行っていないのだろうけど、さて困った。
>>> parser1 = CaboCha.Parser() >>> parser2 = CaboCha.Parser() >>> tree1 = parser1.parse("吾輩は猫である。") >>> tree2 = parser2.parse("吾輩は猫ではない。") >>> tree1.toString(CaboCha.FORMAT_TREE) ' 吾輩は-D\n 猫である。\nEOS\n'
一応回避できることはわかった。これで書くと極めて非python的なプログラミングを強いられるという問題はあるが、たぶんなんとかなる。
ちなみに、ParserのインスタンスがGCに回収されると treeだけ残っててもtoStringできないようです(Segmentation faultを吐いてくれる)。どんな作りになってるのかなんとなくわかってきたけど、率直に言って○○。
def parse(sentences): """ sentencesは一文ずつに区切られた文のリストとして扱う """ trees = [] plist = [] for s in sentences: parser = CaboCha.Parser() trees.append(parser.parse(s)) plist.append(parser)
このようなものを書いてあげれば、後からtreeを使うことができることがわかった。率直に言ってまったく嬉しくない。
問題原因の切り分け
は、できてないです。
- うちの環境固有の問題
- cabocha-pythonの特定のバージョンの問題
- cabocha-python固有の問題
- cabocha固有の問題
とりあえず逃げれることはわかったので、僕はやらない(明言)。
対策
たぶん解析結果のtreeオブジェクトを使いまわそうという発想が間違っていて、cabochaのtreeオブジェクトを使わないで初手でXMLか何かに変換して取り扱うのが楽だと思います。そんなことを強いるバインディングって何よ? って気がしますが。
もう面倒くさいから、JUMAN/KNPに鞍替えしようかなと思う今日この頃。
【python】区切り文字を含めてsplitする
正規表現によるsplitで区切り文字(あるいは区切り文字列)を含めたいときがある。デフォルトでは区切り文字は消える。
たとえば、文を句点で分割する場合。
>>> import re >>> string = "吾輩は猫である。名前はまだない。" >>> re.split("。", string) ['吾輩は猫である', '名前はまだない', '']
実は「区切り文字(列)をリストの要素にする」のか「区切り文字(列)を前の要素に(あるいは後の要素に)くっつける」のかという問題もあるので、一筋縄ではいかない。検索すると前の方ばっかり出てくるので注意。
注意
この記事の内容は一部古いです。おそらく大半の読者が求めているであろうものは追記4にあります。そちらをご覧ください。
区切り文字をリストの要素に入れる
これは検索してたくさん出てくる方。正規表現のグループ化というものを使えば簡単にできる。
>>> re.split("(。)", string) # 半角丸括弧でくくる ['吾輩は猫である', '。', '名前はまだない', '。', '']
今回の例では、それが欲しいんじゃないんだけどなぁ・・・と相成る。
# こうなってほしい(空文字列はご愛嬌) ['吾輩は猫である。', '名前はまだない。', '']
区切り文字を前の要素に付ける。
できるだけ簡単なやり方を考える。
後処理で連結する
真っ先に思いつくのはこの方法。forループで処理する。
>>> lst = [] >>> for elem in re.split("(。)", string): ... if elem != "。": ... lst.append(elem) ... else: ... lst[-1] = lst[-1] + elem ... >>> lst ['吾輩は猫である。', '名前はまだない。', '']
書き方は他にも幾らでもある。この書き方だと、区切り文字(列)が連続したときは連続が途切れるまで連結される。
ただ、これは遅いかもしれない。
正規表現で書く
rubyでやってるページは見つけた。
[Ruby]後読みを使ったsplitで句点(。)を残したまま文に分割 - Qiita
ここの通りにやろうと思ったら、できませんでした・・・。
rubyの正規表現のsplitとは仕様が違うのかなぁ。たぶん『長さゼロの文字列』へのマッチを何らかの方法で書けば同様にできると思われる。どうやるんだろうか、というかできるんだろうか。詳しい人に教えてほしい。
まとめ
正規表現とかよくわからないけど、それに学習コスト投入するのは面倒くさいので騙し騙し適当に処理してプログラム書くことにします・・・。
追記
findallすれば等価のことができた。
>>> re.findall(".*?。", s) ['吾輩は猫である。', '名前はまだない。']
ただし厳密に言えばこれはsplitではない。なのでこうなる。
>>> s2 = "吾輩は猫である。名前はまだない。どこで生まれたかとんと見当がつかぬ" >>> re.findall(".*?。", s2) ['吾輩は猫である。', '名前はまだない。']
邪道だと思うが、
>>> s2 = "吾輩は猫である。名前はまだない。どこで生まれたかとんと見当がつかぬ" >>> re.findall(".*?。|.*$", s2) ['吾輩は猫である。', '名前はまだない。', 'どこで生まれたかとんと見当がつかぬ', '']
こんな方向性で工夫すればなんとかなる可能性はある。正規表現が得意な人なら上手く書く方法を思いつくんだろうなぁ。
追記2
コメントでこういう場合の書き方を教えていただきました。
>>> s2 = "吾輩は猫である。名前はまだない。どこで生まれたかとんと見当がつかぬ" >>> import re >>> re.findall("[^。]+。?",s2) ['吾輩は猫である。', '名前はまだない。', 'どこで生まれたかとんと見当がつかぬ']
やっぱりsplitとは違う気がするけど、実用的には良さそうです。
追記3
※この追記3は以前のバージョンの記述に基づいた内容です。バージョン3.7以降で事情が変わっているので、追記4をご参照ください。
ドキュメントをつらつらと眺めていたら、関連する記述を見つけたのでメモ。
現在、split() は空のパターンマッチでは文字列を分割しません。例えば、次のようになります:
>>> re.split('x*', 'axbc') ['a', 'bc']'x*' は 'a' の前、 'b' と 'c' との間、 'c' の後の 0 個の 'x' にもマッチしますが、現在これらのマッチは無視されます。正しい動作 (空のマッチでも文字列を分割し、['', 'a', 'b', 'c', ''] を返す) は、Python の将来のバージョンで実装されます。これは、後方互換生のない変更であるため、移行期間中は FutureWarning が送出されます。
空の文字列のみとマッチするパターンは、現在文字列を全く分割しません。これは望ましい動作ではないため、Python 3.5 から ValueError が送出されます:
>>> re.split("^$", "foo\n\nbar\n", flags=re.M) Traceback (most recent call last): File "<stdin>", line 1, in <module> ... ValueError: split() requires a non-empty pattern match.
将来的にはできるようになるけど、今のpythonは空のマッチはできないよ、と書いてある。
たとえば、下記の記事のようにすると、
>>> import re >>> re.split("(?<=。)", 'おはよう。こんにちは。こんばんは。') ValueError: split() requires a non-empty pattern match.
こうなるからできない、と。
ただしその下に記述があるように、findall, finditerは空のマッチを含む。finditerなら位置の情報が得られる。
なので、こんな感じで表題の通りの動作は実現できる。
>>> s = 'おはよう。こんにちは。こんばんは。' >>> result = [] >>> before_start = 0 >>> for mobj in re.finditer("(?<=。)", 'おはよう。こんにちは。こんばんは。'): ... result.append(s[before_start:mobj.start()]) ... before_start = mobj.start() ... >>> result ['おはよう。', 'こんにちは。', 'こんばんは。']
これがさえたやり方かどうかはわからないけど、最初にやりたかったことには一番近いのかな・・・? という気がする。
追記4
python3.7から空のマッチに対応し、上のような面倒なことが不要になりました。
バージョン 3.7 で変更: 空文字列にマッチしうるパターンでの分割をサポートするようになりました。
>>> import re >>> re.split("(?<=。)", 'おはよう。こんにちは。こんばんは。') ['おはよう。', 'こんにちは。', 'こんばんは。', '']
ここにたどり着くまでは長い道のりでした。ようやく理想的な結果が得られました。