【python】距離・非類似度行列を計算する
記事概要
非類似度行列(距離行列)の計算方法について説明する。
計算方法
対象データと使う非類似度
とりあえず、データを5つ作る。irisの先頭5要素を抽出する。
from sklearn.datasets import load_iris iris = load_iris() data = iris.data[:5]
5*5の非類似度行列を作りたいというシチュエーションである。
スポンサーリンク
リストとループを使った方法
scipy.spatial.distanceを使うと距離(非類似度)の計算は簡単にできる。
scipy.spatial.distance.pdist — SciPy v1.2.1 Reference Guide
euclideanとcosineを使ってみることにする。
愚直にループを回して行列にしたのが以下のプログラム。
import numpy as np from scipy.spatial.distance import euclidean, cosine from sklearn.datasets import load_iris iris = load_iris() data = iris.data[:5] result = [] for dist in [euclidean, cosine]: tmp1 = [] for v1 in data: tmp2 = [] for v2 in data: tmp2.append(dist(v1, v2)) tmp1.append(tmp2) a = np.array(tmp1) print(a) result.append(a)
実行結果。
[[0. 0.53851648 0.50990195 0.64807407 0.14142136] [0.53851648 0. 0.3 0.33166248 0.60827625] [0.50990195 0.3 0. 0.24494897 0.50990195] [0.64807407 0.33166248 0.24494897 0. 0.64807407] [0.14142136 0.60827625 0.50990195 0.64807407 0. ]] [[0.00000000e+00 1.42083650e-03 1.26527175e-05 8.99393151e-04 2.42323318e-04] [1.42083650e-03 0.00000000e+00 1.20854727e-03 1.20606955e-03 2.75920410e-03] [1.26527175e-05 1.20854727e-03 0.00000000e+00 7.83016618e-04 3.31860741e-04] [8.99393151e-04 1.20606955e-03 7.83016618e-04 0.00000000e+00 1.28295812e-03] [2.42323318e-04 2.75920410e-03 3.31860741e-04 1.28295812e-03 0.00000000e+00]]
基本的にはこれでできるということ。問題点としては、
- 対角成分とその上下は同じなのに二回計算するのは無駄
- オーバーヘッドが気になるかもしれない
- 冗長
ということが主に挙げられるだろう。自分でループの条件を工夫したり速い書き方になるよう努力して対処しても良いが、それすらscipyを使うと簡単にできる。
scipyの関数を使った方法
行列にするところまでscipyで処理することもできる。
やり方は恐らく一つではないが、今回はscipy.spatial.distance.pdistとscipy.spatial.distance.squareformが目についたので使ってみることにする。
pdistは対角成分で分けた行列の半分を計算する。これは一次元配列で結果を返すので、squareformで二次元配列に変換する(squareformは適当にコピーして形を変えるだけで、それほど本質的な処理はしていない)。
from scipy.spatial.distance import pdist, squareform from sklearn.datasets import load_iris iris = load_iris() data = iris.data[:5] for dist in ["euclidean", "cosine"]: print(squareform(pdist(data, metric=dist)))
結果は上と同じなので省略。短く書けるのが素晴らしい。
2019/02/21追記:pdistとsquareformについては以下の記事も御覧ください。
まとめ
できた。
おまけ(非類似度・距離と類似度の関係について)
たまに頭がこんがらがるのでまとめておくと、次のような関係になる。あまり厳密ではないので注意。
- 類似度とは、sim(a, a) = 1となるmetric
- 距離とはdist(a, a) = 0となるmetric
- 非類似度とはdissim(a, a) = 0となるmetric
同じもの同士が0になるか1になるかという観点からだけ見れば、距離と非類似度は同じものである。ただし、非類似度は普通は0~1の値を取るものに対して言うのではないだろうか。
また、0~1の値を取る場合、類似度に変換するにはよく見る次の式が使える。
【python】sklearnライクなデータセットを作る
自作したりネットから拾ってきたデータセットに、sklearnライクなインターフェースがあるとそこそこ便利です。
なので、作る方法について調べました。
とりあえずデータセットを読み込んで型を調べます。
>>> from sklearn.datasets import load_iris >>> iris = load_iris() >>> type(iris) <class 'sklearn.utils.Bunch'>
Bunchという型らしいです。
ではこのBunchを継承したクラスを定義してデータセットのオブジェクトを作るのかというとそうではなく、実はこのBunchというのは「キーにattribとしてアクセスできる辞書のようなもの」であり、コンストラクタに渡したキーと値のセットを内部で作ります。わかりづらいと思いますので例を示すと、
>>> from sklearn.datasets.base import Bunch >>> hoge = Bunch(hogehoge="hoge!") >>> hoge.hogehoge 'hoge!'
こうやって使えるということです。sklearnのソースを読んでも、このような形で使っているので間違いないようです。
https://github.com/scikit-learn/scikit-learn/blob/master/sklearn/datasets/base.py
なので、自作のsklearnライクなデータセットを作りたいということであれば、このBunchをimportしてあげた上で、
Bunch(data=自作したnumpy配列のデータなど, target=自作したnumpy配列のターゲットなど)
こんな形で書けば良いということです。あとはload_***関数を作ってこのBunchのインスタンスをreturnしてやれば完了です。
ちなみに、Bunchのattribはその気になれば外部から上書きすることも可能。実際のdatasetsの実装もそうなっているようです。些か危ない気もしますが、pythonによくある「紳士協定で使え(間違った使い方なんかしないよね)」という奴でしょう。しかし、in-placeで処理される関数をうっかり呼び出したら普通に事故る気が・・・。
>>> from sklearn.datasets import load_iris >>> import numpy as np >>> iris = load_iris() >>> iris.data[:10] array([[5.1, 3.5, 1.4, 0.2], [4.9, 3. , 1.4, 0.2], [4.7, 3.2, 1.3, 0.2], [4.6, 3.1, 1.5, 0.2], [5. , 3.6, 1.4, 0.2], [5.4, 3.9, 1.7, 0.4], [4.6, 3.4, 1.4, 0.3], [5. , 3.4, 1.5, 0.2], [4.4, 2.9, 1.4, 0.2], [4.9, 3.1, 1.5, 0.1]]) >>> np.random.shuffle(iris.data) >>> iris.data[:10] array([[7.7, 2.8, 6.7, 2. ], [5.2, 3.5, 1.5, 0.2], [6.5, 3. , 5.8, 2.2], [5.1, 3.8, 1.5, 0.3], [6.2, 2.8, 4.8, 1.8], [4.4, 2.9, 1.4, 0.2], [4.9, 3. , 1.4, 0.2], [4.3, 3. , 1.1, 0.1], [6.4, 3.1, 5.5, 1.8], [6.8, 3.2, 5.9, 2.3]])
やはり事故るのだった。ちょっと怖いですねぇ。read-onlyにするのはそこまで難しくはないと思うのですが、敢えてそうしていないのか、単に実装がヘボいのか。
追記
公式リファレンス見たら、「Bunchオブジェクトは俺たちが便利に使うために用意しているだけだ、自分のデータセットで同じものを作ろうとはするな、Xとyを定義すりゃいいんだ」と言ってました。
Frequently Asked Questions — scikit-learn 0.22.2 documentation
自作データセットと組み込みのデータセットとでAPIを統一したいシチュエーションでは、ちょっと困りますね。でもまあ、やるなと言っているので、特殊な事情があるとき以外は自重しましょう。
【python】SOMのライブラリSomocluはかなりおすすめ
SOM(Self-organizing maps:自己組織化写像)は割と古めの、データの可視化手法です(それ以外にも使えると思いますが)。
今回はpythonのSOMライブラリSomocluを使ってみたら、けっこう良かったというネタです。
目次
スポンサーリンク
SOMの概要
昨今は深層学習が流行りですが、SOM、自己組織化写像は敢えて言えば単層学習とでも言うべきでしょうか。平面上だったり立体状(まあ理屈の上では何次元でも定義できる)に並べたニューロンにデータをマッピングします。それ以上の説明はwikipediaとか、ググれば色々出てくるページを読んでください。
- wikipedia
- 九州工業大学大学院の先生が書いた読みやすかったページ
http://www.brain.kyutech.ac.jp/~furukawa/data/SOMtext.pdf
- わかりやすい解説
ライブラリがない
SOM、けっこう面白い性質があるみたいなのて使ってみたいのですが、ググってみるとpythonで使えそうなライブラリがとにかくあまり出てきません。
- SOMPY
申し訳ないけど、ちょっと使いづらかった。というかインストールしても挙動が変な感じだった。
GitHub - sevamoo/SOMPY: A Python Library for Self Organizing Map (SOM)
- sompy
日本人の方が実装されたようです。率直に言って「作ってみた」レベルで、実用にはどうかという感じ
自己組織化マップ(SOM)のPythonライブラリsompyを公開しました - 俺とプログラミング
- PyMVPA
多変量解析のためのそれなりに大きいライブラリで、SOMも実装されている。これが使えればよかったのだと思うが、python2系のサポートしかないので没・・・。
Self-organizing Maps — PyMVPA 2.6.1.dev1 documentation
他にも色々あったのですが、割愛。古い手法なので、敢えて作ろうという人がいないのかな・・・。
というか、SOMでググると「実装してみた」系の記事はたくさん出てくるのに、まともに使えるライブラリは出てこないというの、かなり異常というか残念というか・・・。
それでも頑張ってググった
Somocluというのを見つけました。
Introduction — Somoclu 1.7.5 documentation
ウリの部分を適当に訳したり訳さなかったりしつつ抜粋
- OpenMPとCUDAがサポートされていてGPUでも計算できる
- 当然マルチプラットフォームでLinux, macOS, and Windowsでサポートされている
- 「Planar and toroid maps」平面とドーナツみたいな形のSOM両方が作れる
- 「Rectangular and hexagonal grids」四角と六角形がいける
- 「Gaussian or bubble neighborhood functions」近傍の計算を効率化する系のがある
- 「Visualization of maps, including those that were trained outside of Python.」
- マップの初期化にはPCAが使える
すごく良さそう。あと、pythonに依存しないツールでコマンドラインから直接コマンドで叩けます。pythonバインディングもあるよ、という位置づけ。真剣に開発されてる感じです。
使ってみた
とりあえず使ってみました。SOMの可視化結果でよく見るU-matrixという奴を出します。以下のコードで動きました。
# coding: UTF-8 import numpy as np from somoclu import Somoclu from sklearn.datasets import load_iris from sklearn.decomposition import PCA def main(): # データを読み込む dataset = load_iris() X = dataset.data y = dataset.target # SOMに入れる前にPCAして計算コスト削減を測る(iris程度では無駄) pca = PCA(n_components=0.95) X = pca.fit_transform(X) # SOMの定義 n_rows = 16 n_cols = 24 som = Somoclu(n_rows=n_rows, n_columns=n_cols, initialization="pca", verbose=2) # 学習 som.train(data=X, epochs=1000) # U-matrixをファイル出力 som.view_umatrix(labels=y, bestmatches=True, filename="umatrix.png") if __name__ == "__main__": main()
説明不要な感じ。コードも直感的だし、特に不満がないです。
こんな画像が出てきます。

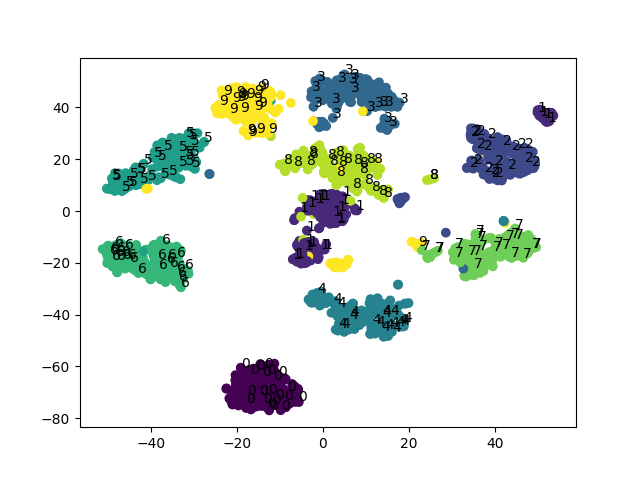
この画像の見方は色の濃淡が重要で、色の明るい部分は相対的に縮尺が縮んでおり、逆に暗い部分は縮尺が相対的に大きい訳です。PCAで可視化した結果を参考に貼っておきます。

紫がラベル0に、緑と黄色が1と2に対応している訳です。SOMを使うと、このようにデータの構造を捉えることができます。
使いやすいし動作もまともだし、Somocluは素晴らしいライブラリです。SOMが必要になったら積極的に使っていきたいところ。
今どきSOMなんか使うの?(蛇足パート)
t-SNEみたいなよくできた手法があるのに今更SOM? と思う方もおられるかと思いますが、SOMはSOMでメリットがあると感じています。
というのは、t-SNEはけっきょくパラメタに依存するし、ミクロな構造を捉えるのは得意でもマクロな構造はどこまで正しいのか? という問題があるからです。
例として、digitsを可視化してみます。
# coding: UTF-8 import numpy as np from sklearn.datasets import load_digits from sklearn.manifold import TSNE from sklearn.decomposition import PCA from somoclu import Somoclu import matplotlib.pyplot as plt def main(): print("loading data") digits = load_digits() pca = PCA(n_components=0.95) pca_data = pca.fit_transform(digits.data) # tsneで可視化 print("tsne") tsne = TSNE() X = tsne.fit_transform(pca_data) fig, ax = plt.subplots() plt.scatter(X[:,0], X[:,1], c=digits.target/10) i = 0 for xy, l in zip(X, digits.target): if i%8 == 0: # 描画されるtextが多いと汚いので省く ax.annotate(l, xy=xy) i += 1 plt.savefig("tsne_digits.png") # somで可視化 print("som") # データを適当に省く sample_index = np.random.choice(X.shape[0], 400, replace=False) sample_X = pca_data[sample_index] sample_y = digits.target[sample_index] # som som = Somoclu(n_rows=30, n_columns=40, initialization="pca") som.train(data=sample_X, epochs=1000) som.view_umatrix(labels=sample_y, bestmatches=True, filename="som_digits.png") if __name__ == "__main__": main()

一見するとt-SNEは同じラベルごとにまとまっていて綺麗なんですが、形の似ている数字が近くに来るのはむしろSOMの方という気もします。0の周りに5,6,9が来るというのは(数字の形を考えると)妥当そうですね。主観的になってしまいますが、SOMも捨てたものではないという気がします。
まとめ
SOMとSomocluは良いのでみんな使おう。
【python】クラス変数のスコープには注意が必要
pythonでクラスを書くとき、一番多用するのは(恐らく)インスタンス変数ですが、クラス変数もたまに使います。
しかし、pythonのクラス変数にはちょっと「クセ」があります。リスト内包表記と組み合わせたときに問題になることが多いようです。
問題の例
リスト内包表記と組み合わせ、他のクラス変数を使いまわしてクラス変数を定義しようとした例です。
class Hoge: x = 5 y = [x for i in range(10)]
なんと以下のエラーを吐きます。
NameError: name 'x' is not defined
こんな人畜無害そうなコードなのに、どうしてなのでしょう。
原因
クラス変数のスコープの取り扱いが原因です。以下のstackoverflowのページによくまとまった説明があります。
Names in class scope are not accessible. Names are resolved in the innermost enclosing function scope. If a class definition occurs in a chain of nested scopes, the resolution process skips class definitions.
The class’s suite is then executed in a new execution frame (see section Naming and binding), using a newly created local namespace and the original global namespace. (Usually, the suite contains only function definitions.) When the class’s suite finishes execution, its execution frame is discarded but its local namespace is saved. [4] A class object is then created using the inheritance list for the base classes and the saved local namespace for the attribute dictionary.
要約すると、クラス変数の中にスコープがネストされた場合、クラス変数の定義されたスコープは読まないよ、ということを言っています。リスト内包表記はスコープを作るので、この問題が起こります。
どうしてこんな仕様になっているんでしょう? 上のリンク先でも貼られているPEP 227のページに解説があります。
PEP 227 -- Statically Nested Scopes | Python.org
Names in class scope are not accessible. Names are resolved in the innermost enclosing function scope. If a class definition occurs in a chain of nested scopes, the resolution process skips class definitions. This rule prevents odd interactions between class attributes and local variable access. If a name binding operation occurs in a class definition, it creates an attribute on the resulting class object. To access this variable in a method, or in a function nested within a method, an attribute reference must be used, either via self or via the class name.
「それができるとメソッド定義で事故るだろーが、親切なpython様が対策してやってるんだよ」と書いてあります。
class Hoge: x = 5 def fuga(self): x = 10 print(x)
色々な事故を招きそうなコードです。こういうことはできない方が妥当で、クラス変数にはHoge.xとしてアクセスするべきです。だけど、この措置がまったく無関係のリスト内包表記にまで影響してしまっています。
じゃあ、こうすれば動くのか?
class Hoge: x = 5 y = [Hoge.x for i in range(10)]
動きません。
NameError: name 'Hoge' is not defined
エラー内容が変わります。Hogeの定義が終わっていない以上、Hoge.xにも当然アクセスできない、ということです。困ったもんですね。
解決策
こういう場合、lambda式による関数呼び出しなどを使って「ネストされたスコープ」にクラス変数を届けてやれ、とリンク先で解説されています。
class Hoge: x = 5 y = (lambda x:[x for i in range(10)])(x)
スコープのネストさえなければ他のクラス変数は使えるし、いったん関数スコープを作ってやれば、あとは普通の名前解決のルールで処理されるので大丈夫な訳です。
これは面倒くさいし、慣れていない人にはラムダ式を直接呼び出しているこのコードは可読性が低いだろうし*1、コードレビューなどで「何やってんのこれ?」とか突っ込まれるくらいならまだ良い方で、これで書いたコードを勝手に他の誰かが直しちゃって大惨事、という事態も想定されます。なので、複数人で管理するコードでは、
- コメントで何をやっているのかしっかり明記する。このページのURLを貼っておくのもおすすめです(爆)
- そもそも使わないで回避策で書く
このような対策が必要になるかと思います。
回避策というのは、要するにリスト内包表記を使わなければ良いので、たとえば今回例として出したコードならこう書けます。
class Hoge: x = 5 y = [x]*10
記述もすっきりするし、どうしてもリスト内包表記を使わないと書けないような処理(そんな処理はほぼない。最悪普通のfor文で書ける)以外では、積極的にリスト内包表記を使う理由はありませんね・・・。
まとめ
- クラス変数のスコープにはクセがあり、リスト内包表記と組み合わせて使うと事故る
- 正面から対策するのは厄介
- 逃げちゃえ
*1:私はpythonワンライナーで遊んできた経験があるので、この手のコードにも慣れてしまってなんとも思いませんが
emacsでpythonを書いているとき「arithmetic error」
tabキーでインデントしようとすると表題の通りのエラーが出て、インデントできない状況になった。
ググって出てきたのはここ。
Indentation not working properly in emacs for python - Stack Overflow
Check the value of python-indent-offset. If it is 0, change it M-x set-variable RET python-indent-offset RET 4 RET.
この対処法で確かに直った。emacsがインデントの設定を推測しようとして、勝手に変になったということらしい。よくわからない。けど、解決すれば良いや。
同じことを繰り返されると面倒なので、init.elにも以下の記述を追加しておく。どうせ空白スペース4つ以外のインデントなんか使わないし。
(add-hook 'python-mode-hook
(lambda () (setq python-indent-offset 4)))
根治できたかどうかはわからないが、とにかく解決はした。
掛け算および割り算するとそれぞれある値になる数字の組を求める
プログラミングにはあまり関係のないテーマだし、中学校レベルの数学がわかればできるネタだが……ちょっと欲しくなったので。
問題
の2つの数(とりあえず正の実数)を考える。
は次の条件を満たす。
が与えられたとき、
を適当に定めたい。
つまり、ディスプレイの画素数と縦横比が決まっているとき、x画素*y画素のxyがそれぞれどうなるかを求めたい、という形の問題。こんなことに数分悩んでしまった。
解く
両方の式を掛けあわせる。
簡単に解けた。
試してみる
pythonで書いてみる。とする。
>>> from math import sqrt >>> def f(c1, c2): ... a = sqrt(c1*c2) ... b = a/c2 ... return a,b ... >>> f(100, 3/4) (8.660254037844387, 11.547005383792516) >>> a, b = f(100, 3/4) >>> a*b 100.00000000000001
上手く行っている。
まとめ
ただの簡単な連立方程式なので解けば良いだけだった。
【python】三項演算子のネストには注意
三項演算子(条件演算子)はpythonの文法でもっとも特徴的な要素かもしれません。
Trueのとき if 条件 else Falseのとき
これには賛否両論がありますが、とにかく便利なことには違いありません。
ただし、ネストして使うときは注意が必要です。
A if condition1 else B if condition2 else C
こういうものを書いた場合、二通りに解釈できますね。
((A if condition1 else B) if condition2 else C) (A if condition1 else (B if condition2 else C))
実行してみるとわかりますが、下の解釈になります。
>>> "A" if True else "B" if False else "C" 'A' >>> "A" if False else "B" if False else "C" 'C'
if condition1 elseの三項演算子を先に認識し、 condition2の方はそのelse項(と言うしかない)とみなしている訳です。まあ、これは自然な仕様でしょう。
ではかっこを付けたときの評価の順番を考えてみましょう。条件演算子では、まず条件が評価されます。それからTrueの場合か、Falseのときのどちらかが評価されます。
なので、かっこで囲った方の優先順位が下がる(後に評価される)ような挙動になります。「かっこでくくると優先順位が上がる(先に評価される)」と捉えていると混乱します。思い通り動かすためにはかっこで囲う必要がある場合がほとんどなのに、それすら思い通り動かないというひどい状態に陥ります。
コツは構文木とか決定木を書くつもりでグルーピングしていくことです。そうすると一応思い通り書けるようになります。
注意が必要だね、という話でした。そもそもそういう事態(三項演算子の多重ネスト)を避けた方が聡明といえば、それまでかもしれませんが、必要に迫られて書くときは注意してください。
【python】数字を1桁ずつに分解
こういう処理がしたいときがある。
i = 2049 lst = [] while i > 0: lst.append(i%10) i //= 10 # 必須 lst.reverse() print(lst) # 結果-> [2, 0, 4, 9]
「もうできたじゃん」という意見もあると思うが、C言語のコードみたいでいかにもpythonicじゃない。
これはこれで良いとして、もっと良い方法がないか考えてみる。
先行研究
同じことを考えた人はたくさんいるようだ。日本語でググって出てきたページを幾つか貼る。
python で 数値を桁別に取得するには - スタック・オーバーフロー
数値を桁ごとにリストに格納する方法,またその逆(Python) - done is better than perfect
Python - python リスト型 変数|teratail
方法としては、上の方法の変形か、strに変換して1桁ずつ読むかのどちらかという感じ。
strに変換するというのは次のようなコード。
[int(i) for c in str(i)]
記述量は少ないが、いかんせん無駄に型変換が走るのはスマートではない。
別の方法1
再帰で書いてみた。
def digit(i, lst=[]): if i > 0: lst.append(i%10) return digit(i//10, lst) else: lst.reverse() return lst print(digit(2049)) # 結果-> [2, 0, 4, 9]
lispだったら綺麗な奴。pythonだと微妙かも。lstを使い回すのが嫌なら、下の書き方もある。
def digit(i): if i > 0: return digit(i//10) + [i%10] else: return []
どうせ末尾再帰最適化なんてされないので、listオブジェクト生成のオーバーヘッド以外にデメリットはない(そのオーバーヘッドがでかかったりするが)。appendを使えばそれすら消せそう。
そして、これができるということは、関数定義だけならワンライナーにもできる。
digit = lambda i:(digit(i//10) + [i%10]) if i > 0 else []
なぜこんなことをするのか? 私の趣味です。
別の方法2
ジェネレータを使って書くこともできると思う。やってない。
別の方法3
思いつかなかった・・・。
まとめ
普通にC言語っぽく書けば良い気がしてきたなぁ・・・。
追記
実行速度を測っていなかったので、計測してみました。
import timeit def f1(i): lst = [] while i > 0: lst.append(i%10) i //= 10 # 必須 lst.reverse() return lst def f2(i): return [int(x) for x in str(i)] print(timeit.timeit(lambda :f1(1))) print(timeit.timeit(lambda :f2(1))) """ => 0.47740281200094614 1.1206639419979183 """ print(timeit.timeit(lambda :f1(1234567890))) print(timeit.timeit(lambda :f2(1234567890))) """ => 2.2151244249980664 3.7194496169977356 """ print(timeit.timeit(lambda :f1(int("1234567890"*10)))) print(timeit.timeit(lambda :f2(int("1234567890"*10)))) """ => 28.711613783001667 25.051117279996106 """
桁数が小さければ割り算で処理する方法が速いが、大きくなるとどこかで逆転する。違いは倍程度には収まるという結果になりました。
まあ、そしたらstrに変換して1桁ずつでも良いかという気もしなくもなく・・・。
【python】in演算子は遅いのか?
はじめに
inの速度は謎である。とりあえず、なんとなく速くはないイメージはあるといえばあるので、ループの中でinを書くとちょっと罪悪感のようなものを感じる。
が、実際のところどうなのだろう?
こちらのページに計算量は出ている。
Pythonistaなら知らないと恥ずかしい計算量のはなし - Qiita
それによると、リストに対するinは平均O(n)で最悪はよくわからない。setに対するinは平均O(1)、最悪O(n)。
つまりリストに対してin演算子を使うと線形探索される。一方、setはhashで実装されていると主張している。まあ、頷ける話ではある。
とはいえ、計算量だけわかってもしょうがない。なので実測することにした。
検証
次のようなベンチマークを考えた。
- range(n)の値を持つコレクションを作り、range(2*n)の値に対してin演算子を実行。トータルの実行時間を測る。
対象にしたコレクション型はlist, set, dict(dictのkey)の3通り。nは100, 200, 400, 600, 800, 1000, 2000, 4000, 6000, 8000, 10000。CPUはintel i7 3540Mで、プログラムはwindows上で動くvmware playerの仮想環境上のubuntuで実行。python 3.5.1。
コードを以下に示す。
# coding: UTF-8 import time import pandas as pd import matplotlib.pyplot as plt def measure(n, mode): if mode == "list": collection = list(range(n)) elif mode == "set": collection = set(range(n)) elif mode == "dkey": collection = dict(zip(range(n), range(n))) else: return None # measure overhead of for t1 = time.time() for i in range(2*n): pass t2 = time.time() overhead = t2 - t1 t1 = time.time() for i in range(2*n): i in collection t2 = time.time() return t2 - t1 - overhead def main(): df = pd.DataFrame([], columns=["n", "list_time", "set_time", "dict_key_time"]) for n in [100, 200, 400, 600, 800, 1000, 2000, 4000, 6000, 8000, 10000]: list_time = measure(n, "list") set_time = measure(n, "set") dkey_time = measure(n, "dkey") s = pd.Series([n, list_time, set_time, dkey_time], index=df.columns) df = df.append(s, ignore_index=True) print(df) df.plot.line(x=["n"]) plt.savefig("result.png") plt.yscale('log') plt.savefig("result_log.png") if __name__ == "__main__": main()
結果
プログラムのテキスト出力を以下に示す。
n list_time set_time dict_key_time 0 100.0 0.000230 0.000004 0.000004 1 200.0 0.000949 0.000008 0.000008 2 400.0 0.003813 0.000044 0.000018 3 600.0 0.008644 0.000030 0.000031 4 800.0 0.015412 0.000040 0.000040 5 1000.0 0.024412 0.000055 0.000068 6 2000.0 0.097047 0.000107 0.000109 7 4000.0 0.402630 0.000255 0.000235 8 6000.0 0.882451 0.000401 0.000443 9 8000.0 1.565103 0.000492 0.000473 10 10000.0 2.448165 0.000576 0.000587
結果のグラフを次の図に示す。

とりあえず、listが劇的に遅い。しかも、リストの大きさに比例するどころか、二次関数みたいな感じになっている(縦軸logの図を見る限り指数関数よりはマシ)。CPUキャッシュなどの影響だと思う。
set, dictのkeyは互角の速度で、listと比べるといかなる要素数においても劇的に速い。1万要素(in演算2万回)で0.000612秒。O(n)と言えるかどうかはぶっちゃけ微妙。hashは衝突回避処理があるので、それが走ると遅くなるのだろうか。それともこれもキャッシュ関係?
結論
listに対するin演算は激遅だった。これは大きいリストに対しては使いたくないと思う。
set, dictのkeyに対するin演算は割と許せる速度なので、使うならこっち。だいたいリストの1/100~1/1000の速度が期待できる。リストでinすると15分かかる処理も1~10秒程度でできるということであり、つまり素晴らしい効能がある。
- listに対してたくさん呼ぶのは地獄
- in演算は無条件で遅い訳ではない。むしろsetなどに対してはそうとう速いので、基本的に使っても大丈夫
これが結論である。なんだか、実際の数字が線形より悪かったり、組み込みのsetが優秀だったりっていうのは、少し意外な感じだった。
cross_val_scoreはもうやめようね。一発で交差検証するにはcross_validateを使う
はじめに
scikit-learnで交差検証を行い、評価指標を算出する方法としては、cross_val_scoreがよくオススメされています。実際、「sklearn 交差検証」みたいな検索キーワードでググるとこの関数がよく出てきます。しかし、この関数は複数の評価指標を算出することができず、一つのスコアしか出力してくれません。
これでどういうとき困るかというと、Accuracy, Precision, Recall, F1をすべて出したい・・・というとき、困ります。基本的にこれらはぜんぶ出して評価するものという考え方のもと検証しようとすると、うまくいかないのです。その辺りを柔軟に制御するために、これまで私は自分で交差検証のコードを書いてきました。
しかし、そんな必要はありませんでした。cross_validateという関数を使えばいいのです。
・・・と、大げさに書いてみましたが、実はこの関数はsklearnのオンラインドキュメントのAPI ReferenceのModel Validationの筆頭に出ています。
API Reference — scikit-learn 0.21.2 documentation
じゃあなんでcross_val_scoreがオススメされるの? というと、cross_validateの方が若干新しいからです。これが使えるのは0.19以降です。それだけの理由ですね。古い情報がそのまま定着してしまって、もっと良いものが出てきてもそれが広まらないというのはよくあることです。
cross_validateの方が何かと柔軟なので、こちらを使いましょう。以下では淡々と説明していきます。
スポンサーリンク
cross_validate
ドキュメントの個別ページはここです。
sklearn.model_selection.cross_validate — scikit-learn 0.21.2 documentation
こんな関数になっています。
sklearn.model_selection.cross_validate(estimator, X, y=None, groups=None, scoring=None, cv=None, n_jobs=1, verbose=0, fit_params=None, pre_dispatch=‘2*n_jobs’, return_train_score=’warn’)
あまりcross_val_scoreと代わり映えしないと言えばしないのですが、scoringに指定できるものがcross_val_scoreとは違います。ちょっと長いですが、当該部分を全文引用します。
scoring : string, callable, list/tuple, dict or None, default: None
A single string (see The scoring parameter: defining model evaluation rules) or a callable (see Defining your scoring strategy from metric functions) to evaluate the predictions on the test set.
For evaluating multiple metrics, either give a list of (unique) strings or a dict with names as keys and callables as values.
NOTE that when using custom scorers, each scorer should return a single value. Metric functions returning a list/array of values can be wrapped into multiple scorers that return one value each.
See Specifying multiple metrics for evaluation for an example.
If None, the estimator’s default scorer (if available) is used.
けっこう凄いことがかいてあります。嬉しいのはlist/tuple, dictが渡せるところです。どう嬉しいかはすぐにわかります。
実例
実際のコードを見ないと良さが伝わらないと思うので、簡単な例を示します。
# coding: UTF-8 from pprint import pprint from sklearn.datasets import load_iris from sklearn.naive_bayes import GaussianNB from sklearn.model_selection import cross_validate, StratifiedKFold def main(): iris = load_iris() gnb = GaussianNB() scoring = {"p": "precision_macro", "r": "recall_macro", "f":"f1_macro"} skf = StratifiedKFold(shuffle=True, random_state=0) scores = cross_validate(gnb, iris.data, iris.target, cv=skf, scoring=scoring) pprint(scores) if __name__ == "__main__": main()
scoresの中身がどうなってるのか? というのがミソの部分で、こうなっています。
{'fit_time': array([0.00096989, 0.00186491, 0.00081563]),
'score_time': array([0.00263953, 0.0025475 , 0.00260162]),
'test_f': array([0.9212963 , 0.98037518, 0.95816993]),
'test_p': array([0.9251462 , 0.98148148, 0.96296296]),
'test_r': array([0.92156863, 0.98039216, 0.95833333]),
'train_f': array([0.95959596, 0.95955882, 0.95096979]),
'train_p': array([0.95959596, 0.96067588, 0.95122655]),
'train_r': array([0.95959596, 0.95959596, 0.95098039])}これを見た瞬間、「もう自分で交差検証を書く必要はない」と私は思いました。欲しい情報はぜんぶ出せます。
・・・といいつつ
不満点はまったくない訳ではなく、
- 各foldでfitさせた分類器がほしい、とか
- 各foldの正解/予測ラベルを生でほしい、とか
いろいろ思うところはあるので、そういうものが必要なときは他のモジュールを探すか(把握しきれてないです、はい)、自分で書くことになります。
それでも普通に数字を出して評価する「だけ」なら一行一発で済む関数があるというのは素晴らしいことです。手間が省けます。素晴らしい。cross_val_scoreはその域には達してないと判断していましたが、cross_validateは使えそうです。
これで簡単に交差検証が書けるようになりました。
まとめ
cross_validateを使おう。
【python】複数の選択肢から確率で選ぶ
おみくじや福引きのようなもの、あるいは強化学習の実装などでタイトルのような「複数のものから確率で選ぶ」処理が必要になることがある。
これについては以前にもこのような記事を書いた。
【python】一定の確率で違う選択をする - 静かなる名辞
この方法でも上手くいくのだが、選択肢が複数になった場合が面倒くさかったり、確率を後から変えるのが大変だったりと、それなりにデメリットがあった。
理想的には、確率のリストなどを渡せばよしなに処理してくれるものがほしい。こんな風に。
何か凄いもの(確率のリスト)
# -> リストのindexを0:15%, 1:35%, 2:20%, 3, 30%の確率で返す
できないと思っていたが、numpyでできることが判明した。ただしversion 1.7.0以上が必要。
numpy.random.choice — NumPy v1.15 Manual
たとえば、a,b,c,dを[0.15, 0.35, 0.2, 0.3]の確率で選びたい場合、単にこうすれば良い。
np.random.choice(["a", "b", "c", "d"], p=[0.15, 0.35, 0.2, 0.3]) # -> "a", "b", "c", "d"のいずれかが確率で返る
想像以上に簡単だ・・・。
インデックスを返させることもできる。
np.random.choice(4, p=[0.15, 0.35, 0.2, 0.3]) # -> 0, 1, 2, 3のいずれかが確率で返る
0,1,2,3を選んでくれる。素晴らしい。
更に、複数の結果を吐かせることもできる。
np.random.choice(4, size=10, p=[0.15, 0.35, 0.2, 0.3]) # -> 一例:array([3, 2, 3, 3, 1, 3, 2, 1, 1, 0])
更に更に、replaceというオプションがある。これは重複するかどうかを指定する(ふくびきで出た玉を一々中に戻すかどうかに相当)。defaultはTrueで、玉を中に戻すことに相当する。Falseにすると次のようなことができる。
np.random.choice(5, 5, replace=False) # -> array([3, 0, 4, 1, 2])
これはランダムサンプリングするときに使えるだろう。ただし、同時にpオプションを指定すると上手く動かない。次のエラーが出る。
ValueError: Cannot take a larger sample than population when 'replace=False'
何はともあれ、こんな便利なものがあることは知らなかった。普段numpyのドキュメントを積極的に読もうと思わないので、こういうことに気づかないパターンが多い。
機会があったら使っていこう・・・。
【python】RandomTreesEmbeddingを試す(1)
RandomTreesEmbeddingはsklearnにたくさんある謎クラスの一つ*1。
たぶんスパースコーディングに決定木を使いましょうね~系の奴なんだと思う。
ドキュメントを読むと、なんとなく雰囲気はわかる。
sklearn.ensemble.RandomTreesEmbedding — scikit-learn 0.20.1 documentation
あー、なるほど、決定木を作った後、木のパスを符号にしてるのね(わかってない。でもハフマン符号みたいなものかな、という見当はつく)。
とりあえずどんなものか試してみる。というかこの記事は本当に試してみただけ。
なにはともあれirisから
いつも通り分類問題の前処理をやらせてみよう。
プログラム。
# coding: UTF-8 from sklearn.datasets import load_iris, load_digits from sklearn.ensemble import RandomTreesEmbedding as RTE,\ RandomForestClassifier as RFC from sklearn.naive_bayes import GaussianNB from sklearn.model_selection import cross_validate def test_func(dataset, clf, embed=True): if embed: rte = RTE(n_estimators=100, sparse_output=False, n_jobs=-1) X = rte.fit_transform(dataset.data) else: X = dataset.data print(X.shape) scoring = {"p": "precision_macro", "r": "recall_macro", "f":"f1_macro"} scores = cross_validate(clf, X, dataset.target, scoring=scoring, return_train_score=False, n_jobs=-1) print("p:{0:.5f} r:{1:.5f} f:{2:.5f}".format( scores["test_p"].mean(), scores["test_r"].mean(), scores["test_f"].mean())) def main(): iris = load_iris() digits = load_digits() gnb = GaussianNB() rfc = RFC(n_estimators=1000, n_jobs=-1) print("iris") test_func(iris, gnb, embed=False) test_func(iris, rfc, embed=False) test_func(iris, gnb, embed=True) test_func(iris, rfc, embed=True) print("digits") test_func(digits, gnb, embed=False) test_func(digits, rfc, embed=False) test_func(digits, gnb, embed=True) test_func(digits, rfc, embed=True) if __name__ == "__main__": main()
結果
iris (150, 4) p:0.93611 r:0.93423 f:0.93411 (150, 4) p:0.96768 r:0.96732 f:0.96731 (150, 2155) p:0.96234 r:0.96038 f:0.96027 (150, 2189) p:0.96234 r:0.96038 f:0.96027 digits (1797, 64) p:0.84221 r:0.81910 f:0.82039 (1797, 64) p:0.94671 r:0.94478 f:0.94473 (1797, 2503) p:0.90086 r:0.89582 f:0.89597 (1797, 2488) p:0.93643 r:0.93373 f:0.93403
まあ思った通り、4次元から2000次元以上に張られてしまう奴だった。ナイーブベイズだと分類精度に恩恵があるっぽいが、ランダムフォレストで効く訳もなく。
ちなみに、木の数を増やすと次元数はだいたい比例して増える。おっかない。まあ、逆に言えば木の数で適当な次元数に調整できるということでもあるのだが(他の決定木のパラメタでもできる)。
まとめ
本来の使い方じゃないと思うので、そのうちちゃんとした記事を書きます・・・。
たぶん画像処理とかの問題を解くのに使えるんだと思う。
*1:ひどい言い方だけど、「僕にとって謎」という意味である。予防線張っとく
【python】sklearnのPCAでloading(主成分負荷量)を計算する
PCA(主成分分析)のloading*1がほしいときがあります。
sklearnでは一発では出ません。
ドキュメントはここ。
sklearn.decomposition.PCA — scikit-learn 0.21.2 documentation
目次
PCA.components_は確かにあるけど・・・
結論から先に言うと、PCA.components_はノルム1の固有ベクトルです。ノルムを測ってみましょう。
>>> import numpy as np >>> from sklearn.datasets import load_iris >>> from sklearn.decomposition import PCA >>> iris = load_iris() >>> pca = PCA(n_components=2) >>> pca.fit(iris.data) PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False) >>> pca.components_ array([[ 0.36158968, -0.08226889, 0.85657211, 0.35884393], [ 0.65653988, 0.72971237, -0.1757674 , -0.07470647]]) >>> np.linalg.norm(pca.components_, axis=1) array([1., 1.])
まあ、loadingも固有ベクトルには違いないのですが、ノルムを整えてやる必要があります。
loadingを計算しよう
教科書などによく書いてあることですが、第主成分に対応する元の変数のloadingは次の式で出せます。
は固有値。 eigenvectorは固有ベクトルで、元の変数の数だけ次元がありますから、これで良いわけです(雑な説明ですが・・・)。
ということで、pythonで同様にやってみましょう。固有値はexplained_varianceに入っています。
>>> pca.components_*np.c_[np.sqrt(pca.explained_variance_)] # 縦ベクトルに変換する必要がある array([[ 0.74322652, -0.16909891, 1.76063406, 0.73758279], [ 0.32313741, 0.35915163, -0.08650963, -0.03676921]])
できました。これがloadingです。・・・と思ったけど、1を超えちゃってますね。なんてこった。
罠だった
固有値は分散なので、データのスケールに依存します。
とりあえず入力をスケーリングしてみよう。上の式は相関行列から行くときのものでした。なのでこれで平気なはず。
>>> from sklearn.preprocessing import StandardScaler as SS >>> ss = SS() >>> data = ss.fit_transform(iris.data) >>> pca.fit(data) PCA(copy=True, iterated_power='auto', n_components=2, random_state=None, svd_solver='auto', tol=0.0, whiten=False) >>> pca.components_*np.c_[np.sqrt(pca.explained_variance_)] array([[ 0.89421016, -0.45081822, 0.99500666, 0.96822861], [ 0.35854928, 0.89132754, 0.02031465, 0.06299656]])
1を超えなくなくてめでたし、ということよりも、数字が変わったことの方が問題で、これで本当に正しいのかという疑念が生じてきました。
確認のために元の特徴と主成分の相関係数を直接測ってみます。
>>> X = pca.fit_transform(data) >>> np.corrcoef(np.hstack([iris.data, X]), rowvar=False) array([[ 1.00000000e+00, -1.09369250e-01, 8.71754157e-01, 8.17953633e-01, 8.91224479e-01, 3.57352114e-01], [-1.09369250e-01, 1.00000000e+00, -4.20516096e-01, -3.56544090e-01, -4.49312976e-01, 8.88351481e-01], [ 8.71754157e-01, -4.20516096e-01, 1.00000000e+00, 9.62757097e-01, 9.91684422e-01, 2.02468206e-02], [ 8.17953633e-01, -3.56544090e-01, 9.62757097e-01, 1.00000000e+00, 9.64995787e-01, 6.27862218e-02], [ 8.91224479e-01, -4.49312976e-01, 9.91684422e-01, 9.64995787e-01, 1.00000000e+00, 2.08904471e-17], [ 3.57352114e-01, 8.88351481e-01, 2.02468206e-02, 6.27862218e-02, 2.08904471e-17, 1.00000000e+00]])
下の二行の4列目までを見てください。微妙に誤差があるような気はしますが(小数点以下3桁でずれてきてるので微妙ってほど微妙でもないが)、たぶん同じ数字になっています。
微妙な誤差については、丸め誤差などが累積した、実は計算間違ってる、といった理由が考えられます。前者ならまだ許せるけど、後者はやだな・・・。
共分散行列のときはどうするのか
どうするんだろうね・・・。
2019/06/22追記
手順は増えますが、スケールを考慮すれば同様に行えるようです。
出典:http://manabukano.brilliant-future.net/document/text-PCA.pdf p.10
loadingを使うと何が良いのか
相関係数なので、「どれくらい効いてるか」がよくわかります。よくある0.3以下なら~とか0.7以上なら~という論法ができます。それだけといえばそれだけ。
このように取扱が大変なので、固有ベクトルのまま解釈した方が楽かもという気がしてきました。各主成分の寄与率はexplained_variance_ratio_で得られる訳だし、寄与率の大きい軸の固有ベクトルの大きい次元を見ればどんな感じかはわかるし・・・。
でもまあ、一応(入力をスケーリングすれば)大体出るということはわかったので、これでよしとします。
共分散行列でやるときのやり方は、どなたか詳しい方に教えて頂けると助かります。
*1:主成分負荷量、あるいは因子負荷量とも(なぜか)言われますが、この記事ではloadingで通します。けっきょくヘタに和訳しないのがいちばんわかりやすい
【python】sklearnで因子分析を試す
pythonで因子分析をやる人はあまりいないようだが、sklearnにはしっかりモデルが存在している。ついさっき気づいた。
sklearn.decomposition.FactorAnalysis — scikit-learn 0.20.1 documentation
因子分析自体は前からどんなものなのか興味があり、かといってググるとRだったりSPSSだったりばっかり出てきて辟易していたのだが、sklearnにあると都合が良い。さっそく使ってみよう。
目次
とりあえずirisをプロットする
私だけでも何十回もやってきた、世界中では何万回とやられてきたirisの二次元可視化をやってみる。
次のようなコードを書いた。
# coding: UTF-8 from copy import deepcopy from sklearn.datasets import load_iris from sklearn.preprocessing import StandardScaler from sklearn.decomposition import PCA, FactorAnalysis as FA from sklearn.pipeline import Pipeline import matplotlib.pyplot as plt def decomp_and_plot(dataset, model, file_name): X = model.fit_transform(dataset.data) plt.figure() plt.scatter(X[:,0], X[:,1], c=dataset.target/len(dataset.target_names)) plt.savefig(file_name) def main(): iris = load_iris() ss = StandardScaler() pca = PCA(n_components=2) pl = Pipeline([("scaler", ss), ("pca", deepcopy(pca))]) fa = FA(n_components=2, max_iter=5000) decomp_and_plot(iris, pca, "pca_plt.png") decomp_and_plot(iris, pl, "spca_plt.png") decomp_and_plot(iris, fa, "fa_plt.png") if __name__ == "__main__": main()
PCA、変数をスケーリングしたPCA(相関行列を使うことと等価)、因子分析でそれぞれplotしてみる。
結果はこれ。



とりあえずcomponentsを見る
次のようなmain2を作り、実行した。
def main2(): iris = load_iris() print(iris.feature_names) print("pca") pca = PCA(n_components=2) pca.fit(iris.data) print(pca.components_) print("fa") fa = FA(n_components=2, max_iter=5000) fa.fit(iris.data) print(fa.components_)
結果
['sepal length (cm)', 'sepal width (cm)', 'petal length (cm)', 'petal width (cm)'] pca [[ 0.36158968 -0.08226889 0.85657211 0.35884393] [ 0.65653988 0.72971237 -0.1757674 -0.07470647]] fa [[ 0.72577591 -0.17754023 1.75733754 0.73196365] [-0.37036948 -0.24060118 0.02793388 0.04121372]]
プロット結果から予想される通り、両者のcomponentsはよく似通っている。
これがloadingなのかどうかはぶっちゃけよくわからないのだが(というか1を超えてくる時点でたぶん違うのだろうが)、とりあえずloadingだと思って解釈する。
第一因子は花弁の長さと幅、がく片の長さに対応しているので花の大きさに対応しているっぽい。花の大きさとがく片の幅はなぜか若干反比例する。
第二因子は花弁に関する係数が小さいので、がく片の大きさを表す因子と言って良さそうである。
こんなところか。
使えることはわかった
だから何? って言われると、正直答えに窮しますが・・・とにかく使えます。主成分分析で良いじゃんと言われたら何も言い返せません。
【python】改行せずに代入文と等価のことをする
pythonワンライナーを書く上で障害になるのは、代入文の存在である。
代入は関数ではなく文なので、素直に書くと一行を消費してしまうし、lambdaやコレクション型の中にも入れられない。
よく知られた対策としては、グローバル変数テーブルを直接書き換えるという大技がある。
>>> globals().__setitem__("hoge", 1) >>> hoge 1
でも__setitem__とか呼ぶのは、はっきり言ってキモい。それに、ローカル変数は書き換えられないという問題もある。もっと普通のpythonの枠内でpythonワンライナーを書きたい。
長らくこの問題に悩んでいたが、ついに等価の表現を発見した。リストならアンダーバー付きのメソッドを呼ばなくても行ける。
>>> l = [1] >>> (l.pop(0), l.append(2))[-1] >>> l [2]
ワンライナーとして変数辞書と同様に使える表現を考えてみる。lispのalistを参考にして作る。
(lambda setalist, getalist, alist: # 環境のalistとアクセス用関数を定義(lispのletと同様) ((setalist("hoge", 1, alist), # 以下main print(getalist("hoge", alist)), setalist("hoge", 2, alist), print(getalist("hoge", alist)), setalist("fuga", "fuga", alist), print(getalist("fuga", alist)), print(getalist("hoge", alist)), setalist("lst", [], alist), getalist("lst", alist).extend([1,2,3,4,5]), print(getalist("lst", alist)[0:2]) # mainここまで ,)))( # ここから一番外のlambdaの引数 (lambda key, value, alist: # setalistの実体 (lambda search_result: ((alist.pop(search_result[0]) if search_result != [] else None), alist.append((key, value)), value)[-1])([i for (i, (k,v)) in enumerate(alist) if k==key])), (lambda key, alist: # getalistの実体 (lambda tmp:tmp[0] if tmp != [] else None)( [e[1] for e in alist if key == e[0]])), [] # alistの実体 )
出力
1 2 fuga 2 [1, 2]
ほぼ手続き型ライクに使える。ただし、リストの要素などには直接代入できないので、そういったことに気を配る必要があるが(リストの要素に代入するだけならinsertしてpopすれば良いので関数で書けそう)。
もう少し頑張れば、可読性の高い(アンダーバー付きのメソッドを呼ばなくて良い)pythonワンライナーを書けそうだ。
