【python】SOMのライブラリSomocluはかなりおすすめ
SOM(Self-organizing maps:自己組織化写像)は割と古めの、データの可視化手法です(それ以外にも使えると思いますが)。
今回はpythonのSOMライブラリSomocluを使ってみたら、けっこう良かったというネタです。
目次
スポンサーリンク
SOMの概要
昨今は深層学習が流行りですが、SOM、自己組織化写像は敢えて言えば単層学習とでも言うべきでしょうか。平面上だったり立体状(まあ理屈の上では何次元でも定義できる)に並べたニューロンにデータをマッピングします。それ以上の説明はwikipediaとか、ググれば色々出てくるページを読んでください。
- wikipedia
- 九州工業大学大学院の先生が書いた読みやすかったページ
http://www.brain.kyutech.ac.jp/~furukawa/data/SOMtext.pdf
- わかりやすい解説
ライブラリがない
SOM、けっこう面白い性質があるみたいなのて使ってみたいのですが、ググってみるとpythonで使えそうなライブラリがとにかくあまり出てきません。
- SOMPY
申し訳ないけど、ちょっと使いづらかった。というかインストールしても挙動が変な感じだった。
GitHub - sevamoo/SOMPY: A Python Library for Self Organizing Map (SOM)
- sompy
日本人の方が実装されたようです。率直に言って「作ってみた」レベルで、実用にはどうかという感じ
自己組織化マップ(SOM)のPythonライブラリsompyを公開しました - 俺とプログラミング
- PyMVPA
多変量解析のためのそれなりに大きいライブラリで、SOMも実装されている。これが使えればよかったのだと思うが、python2系のサポートしかないので没・・・。
Self-organizing Maps — PyMVPA 2.6.1.dev1 documentation
他にも色々あったのですが、割愛。古い手法なので、敢えて作ろうという人がいないのかな・・・。
というか、SOMでググると「実装してみた」系の記事はたくさん出てくるのに、まともに使えるライブラリは出てこないというの、かなり異常というか残念というか・・・。
それでも頑張ってググった
Somocluというのを見つけました。
Introduction — Somoclu 1.7.5 documentation
ウリの部分を適当に訳したり訳さなかったりしつつ抜粋
- OpenMPとCUDAがサポートされていてGPUでも計算できる
- 当然マルチプラットフォームでLinux, macOS, and Windowsでサポートされている
- 「Planar and toroid maps」平面とドーナツみたいな形のSOM両方が作れる
- 「Rectangular and hexagonal grids」四角と六角形がいける
- 「Gaussian or bubble neighborhood functions」近傍の計算を効率化する系のがある
- 「Visualization of maps, including those that were trained outside of Python.」
- マップの初期化にはPCAが使える
すごく良さそう。あと、pythonに依存しないツールでコマンドラインから直接コマンドで叩けます。pythonバインディングもあるよ、という位置づけ。真剣に開発されてる感じです。
使ってみた
とりあえず使ってみました。SOMの可視化結果でよく見るU-matrixという奴を出します。以下のコードで動きました。
# coding: UTF-8 import numpy as np from somoclu import Somoclu from sklearn.datasets import load_iris from sklearn.decomposition import PCA def main(): # データを読み込む dataset = load_iris() X = dataset.data y = dataset.target # SOMに入れる前にPCAして計算コスト削減を測る(iris程度では無駄) pca = PCA(n_components=0.95) X = pca.fit_transform(X) # SOMの定義 n_rows = 16 n_cols = 24 som = Somoclu(n_rows=n_rows, n_columns=n_cols, initialization="pca", verbose=2) # 学習 som.train(data=X, epochs=1000) # U-matrixをファイル出力 som.view_umatrix(labels=y, bestmatches=True, filename="umatrix.png") if __name__ == "__main__": main()
説明不要な感じ。コードも直感的だし、特に不満がないです。
こんな画像が出てきます。

この画像の見方は色の濃淡が重要で、色の明るい部分は相対的に縮尺が縮んでおり、逆に暗い部分は縮尺が相対的に大きい訳です。PCAで可視化した結果を参考に貼っておきます。

紫がラベル0に、緑と黄色が1と2に対応している訳です。SOMを使うと、このようにデータの構造を捉えることができます。
使いやすいし動作もまともだし、Somocluは素晴らしいライブラリです。SOMが必要になったら積極的に使っていきたいところ。
今どきSOMなんか使うの?(蛇足パート)
t-SNEみたいなよくできた手法があるのに今更SOM? と思う方もおられるかと思いますが、SOMはSOMでメリットがあると感じています。
というのは、t-SNEはけっきょくパラメタに依存するし、ミクロな構造を捉えるのは得意でもマクロな構造はどこまで正しいのか? という問題があるからです。
例として、digitsを可視化してみます。
# coding: UTF-8 import numpy as np from sklearn.datasets import load_digits from sklearn.manifold import TSNE from sklearn.decomposition import PCA from somoclu import Somoclu import matplotlib.pyplot as plt def main(): print("loading data") digits = load_digits() pca = PCA(n_components=0.95) pca_data = pca.fit_transform(digits.data) # tsneで可視化 print("tsne") tsne = TSNE() X = tsne.fit_transform(pca_data) fig, ax = plt.subplots() plt.scatter(X[:,0], X[:,1], c=digits.target/10) i = 0 for xy, l in zip(X, digits.target): if i%8 == 0: # 描画されるtextが多いと汚いので省く ax.annotate(l, xy=xy) i += 1 plt.savefig("tsne_digits.png") # somで可視化 print("som") # データを適当に省く sample_index = np.random.choice(X.shape[0], 400, replace=False) sample_X = pca_data[sample_index] sample_y = digits.target[sample_index] # som som = Somoclu(n_rows=30, n_columns=40, initialization="pca") som.train(data=sample_X, epochs=1000) som.view_umatrix(labels=sample_y, bestmatches=True, filename="som_digits.png") if __name__ == "__main__": main()
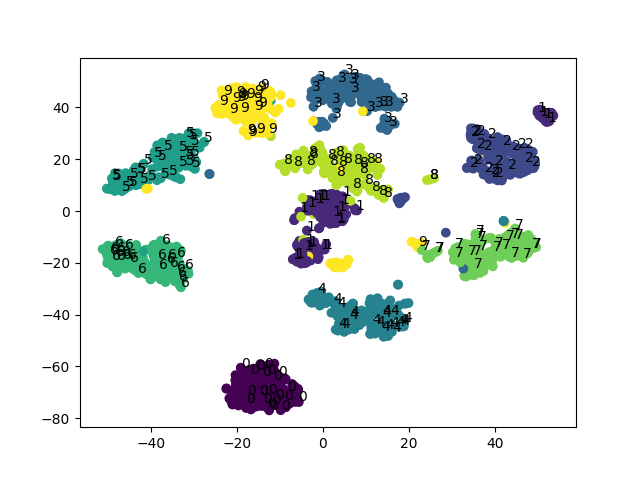

一見するとt-SNEは同じラベルごとにまとまっていて綺麗なんですが、形の似ている数字が近くに来るのはむしろSOMの方という気もします。0の周りに5,6,9が来るというのは(数字の形を考えると)妥当そうですね。主観的になってしまいますが、SOMも捨てたものではないという気がします。
まとめ
SOMとSomocluは良いのでみんな使おう。