【python】seabornで棒グラフを信頼区間付きで描く
はじめに
信頼区間付きの棒グラフはよく見かけます。複数グループの差が優位かどうかという議論に向いているからです。
他のツールだと割と簡単に描けるグラフだったりするのですが、Pythonでやろうとするとmatplotlibは勝手に信頼区間を計算してくれません。信頼区間を計算してからエラーバーを出して……といったノウハウが紹介されていますが、正直面倒くさいです。
そこでseabornを使います。こちらは何もしなくても信頼区間を出してくれるタイプのライブラリです。ただし、使い勝手には癖があります。
リファレンス通りに使ってみる
凝ったことをする意味はあまりないので、まずはリファレンス通りに使います。
seaborn.barplot — seaborn 0.9.0 documentation
リファレンスの記述を多少補ったのが以下のコードです。tipsデータセットでやっているようです。
import seaborn as sns import matplotlib.pyplot as plt tips = sns.load_dataset("tips") print(tips.head()) """ => total_bill tip sex smoker day time size 0 16.99 1.01 Female No Sun Dinner 2 1 10.34 1.66 Male No Sun Dinner 3 2 21.01 3.50 Male No Sun Dinner 3 3 23.68 3.31 Male No Sun Dinner 2 4 24.59 3.61 Female No Sun Dinner 4 """ sns.barplot(x="day", y="total_bill", data=tips) plt.savefig("result.png")
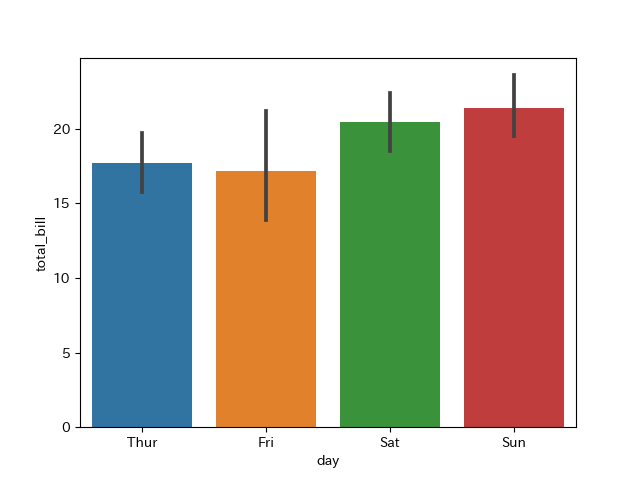
折れ線グラフのときと使い方はほとんど変わりません。というか、見え方が違うだけのようにも見えます(折れ線の方だとx軸の位置は指定できますが)。
【python】seabornで折れ線グラフを信頼区間付きで描く - 静かなる名辞
なお、エラーバーの形式はいじれます。普通は傘がほしいと思うので、capsizeを指定します。また、デフォルトでは少しエラーバーが濃すぎる気がするので、errwidthで調整するとよさげです。
適当に調整したのが下のものです。
sns.barplot(x="day", y="total_bill", data=tips, capsize=0.1, errwidth=1.2)

もともとDataFrameになっていない任意のデータで行う
例によって例のごとく、seabornの開発方針はpandasべったりなので、どんな形式・内容のデータであっても一回DataFrameに突っ込んでからやった方が良さそうです。ただ、受け付けてくれる形式が少し特殊なので、変換方法を覚えておいた方が得です。
普通のデータはこんな感じで定義したいことが多いと思います。棒ごとに配列があるイメージですね。
df = pd.DataFrame({"A": [11, 12, 11, 10, 10, 13, 11, 10, 15, 11, 10],
"B": [20, 21, 20, 21, 20, 21, 20, 21, 20, 21, 20]})
meltを使って縦持ちに変換すると、seabornと親和性の高いフォーマットになります。結論から言うと、こう書くと良いでしょう。
import pandas as pd import seaborn as sns import matplotlib.pyplot as plt df = pd.DataFrame({"A": [11, 12, 11, 10, 10, 13, 11, 10, 15, 11, 10], "B": [20, 21, 20, 21, 20, 21, 20, 21, 20, 21, 20]}) sns.barplot(x="class", y="data", data=df.melt(var_name="class", value_name="data"), capsize=0.1, errwidth=1.2) plt.savefig("result.png")
これでちゃんとABごとに集計してくれます。ちなみにmeltした結果はこうなります。
class data 0 A 11 1 A 12 2 A 11 3 A 10 4 A 10 5 A 13 6 A 11 7 A 10 8 A 15 9 A 11 10 A 10 11 B 20 12 B 21 13 B 20 14 B 21 15 B 20 16 B 21 17 B 20 18 B 21 19 B 20 20 B 21 21 B 20
まとめ
データを縦持ちに変換するところでひと手間余計にかかりますが、matplotlibでゼロからやるよりは楽だし、間違いも少なさそうなので良いと思います。
Python対話的インタプリタでアンダースコアが便利(誰も知らない機能)
概要
対話モードだとアンダースコアの変数が自動的にできています。最後に評価した結果が入るようです。
>>> 1 + 2 3 >>> _ 3
これはチュートリアルに書いてあったのですが、他の入門記事で触れられているのを見た記憶はまったくありません。私自身も、チュートリアルの「Pythonを電卓として使う」はなんとなく読み流していたので気づいていませんでした。
対話モードでは、最後に表示された結果は変数 _ に代入されます。このことを利用すると、Python を電卓として使うときに、計算を連続して行う作業が多少楽になります。
3. 形式ばらない Python の紹介 — Python 3.8.0 ドキュメント
面白いですね。
性質を調べる
あくまでも「最後に評価された式の値」を保持するものなので、標準出力に吐いた結果は反映されません。また、関数呼び出しでも結果は反映されますが、返り値のない関数を呼び出してもNoneになることはないようです。
>>> 4 + 5 9 >>> _ 9 >>> pow(4, 2) 16 >>> _ 16 >>> print("hoge", "fuga") hoge fuga >>> _ 16
ドキュメントを調べる
こんな簡単なものなのですが、ちゃんとしたドキュメントの記述を探そうとすると検索性が悪くて一苦労です。けっきょく正攻法で探すのは諦め、たぶん英語版スタックオーバーフローあたりの質問が出てくるだろうと思って「python interactive shell underscore」でgoogle検索したら、案の定出てきました。
sys.displayhookというのがそれに該当する関数だそうです。
sys --- システムパラメータと関数 — Python 3.8.0 ドキュメント
性質はなかなか面白そうですが、まるごと引用するには少し長かったのでドキュメントをじっくり読んでください。
Jupyterでもできる
あいつも対話環境の端くれならできるかもしれないと思って試したら、できました。

正確な挙動は確認していませんが、試した範囲ではセルの評価が終わった瞬間の結果が代入されているようです。一つのセルで、
10 + 20 _
とやっても望む結果は得られないということです。
まとめ
割と便利。たのしい。
【python】seabornで折れ線グラフを信頼区間付きで描く
はじめに
信頼区間というとなんとなく棒グラフにつけるものという印象がありますが、折れ線グラフでも計算すること自体はたやすくて、しかもけっこうかっこいいグラフになります。
大抵は別に無くても良いのですが、たまに信頼区間が出ていると便利なときがあります(有意な差があるかどうかを視覚的に示せます)。
seabornでできるのでやってみます。
seaborn.lineplot — seaborn 0.9.0 documentation
DataFrameを入力して使う
リファレンスではこの方法が推されています。DataFrameがあるのであれば簡単にできます。
公式の例をパクってきたような感じの実行例を以下に示します。
import seaborn as sns import matplotlib.pyplot as plt fmri = sns.load_dataset("fmri") print(fmri.head()) # どんなデータなのか見ておく """ => subject timepoint event region signal 0 s13 18 stim parietal -0.017552 1 s5 14 stim parietal -0.080883 2 s12 18 stim parietal -0.081033 3 s11 18 stim parietal -0.046134 4 s10 18 stim parietal -0.037970 """ sns.lineplot(x="timepoint", y="signal", data=fmri) plt.savefig("result.png")

何をやっているのかと言うと、timepointで集計してsignalの信頼区間(デフォルトは95%)を計算しています。たったこれだけの指定でこれだけのことをしてくれるので、便利なものです。
裏を返せば、同じx軸の値に対して対応するy軸の値が複数ないと信頼区間は出ません(当たり前のことですが)。
面倒くさいのでただの配列で計算できるようにする
seabornのリファレンスではDataFrameを入力にして使う方法がやたら推されていますが、私はpandasは扱うのが面倒くさいので、numpy配列ですませたいと思うタイプの人間です。
その場合のやり方ですが、まず上に挙げた例は、以下のように書いても同じ結果が得られます。
sns.lineplot(x=fmri["timepoint"], y=fmri["signal"])
性質はよくわかったので、numpyで作ったサイン波でやってみます。
import numpy as np import seaborn as sns import matplotlib.pyplot as plt x = np.repeat(np.linspace(0, 10, 20), 20) # たとえばnp.repeat(np.arange(5), 2)は # array([0, 0, 1, 1, 2, 2, 3, 3, 4, 4])になる性質があります y = np.sin(x) + np.random.normal(size=x.shape) sns.lineplot(x=x, y=y) plt.savefig("result.png")

微妙な使い勝手ですが(どちらかといえば外側でgroupbyした結果で渡したいんだけどな、とか)、使えるには使えます。
ちなみに上のグラフを見て気づいた方もいるかと思いますが、基本的にはxの値でグルーピングしたあとそれぞれのxに対応する信頼区間を計算しているだけのようです。点の信頼区間同士も線形に折れ線で繋がれます(ので、点と点の間で妥当なことになっている保障はないと考えた方が無難です)。
まとめ
折れ線グラフで信頼区間を議論することはあまりないかもしれませんが、それでもたま~に便利なときがあるので、覚えておいて損はないです。
ランダムフォレストを使うなら変数選択はしなくてもいいのか?
はじめに
表題の通りの話をたまに聞きます。「ランダムフォレストは内部で変数選択を行う。なので変数選択は必要ない」という主張です。
しかし個人的には、それはあくまでも
- 他の手法*1と比べれば変数選択しなかった場合の悪影響が少ない
ということであって、ランダムフォレストであっても変数選択した方が良いんじゃ? ということを昔からずっと思っていました。
検証してみます。
思考実験
実際に検証する前に思考実験を行います。
まずパターンA(変数選択なし)とパターンB(変数選択あり)の2通りを考えます。
- パターンA
有効な変数:10個
無効な変数:90個
- パターンB
有効な変数:10個
のみ(無効な変数なし)
ランダムフォレストの弱分類器では、元々の変数の数の平方根くらいの数の変数を使うのが一般的です。そうすると、
- パターンAの場合
弱分類器で使う変数は10個。うち有効なもの(の期待値)は1個。
- パターンBの場合
弱分類器で使う変数は3個(切り捨てたとする)。うち有効なものは3個。
となり、どう見てもパターンBの方が良い結果を生みそうです。期待される結果としては、
- 各弱分類器の性能が上がることで、全体の性能が向上
- 少ない木の本数で済む
- 過学習しづらくなる
などがあり、いいことずくめです。
実験
データセットや条件を変えてやってみたら、変数選択が効くデータや条件、そうでもないデータや条件がいろいろ出てきてしまいました。
要するにケースバイケースなのですが、結果がわかりやすかったやつだけ見せることにします(いい加減)。
やったこと
- データセットはscikit-learnのdigits
- 元々の特徴量ベクトルは64次元だが、ノイズを128次元足して192次元にした
- SelectPercentileで特徴選択して10%ずつ増やしながら見ていく
- 分類器はランダムフォレスト
- 訓練データとテストデータに半分ずつ分けて評価した
sklearn.feature_selection.SelectPercentile — scikit-learn 0.21.3 documentation
コード
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import load_digits from sklearn.model_selection import train_test_split from sklearn.feature_selection import SelectPercentile from sklearn.ensemble import RandomForestClassifier from sklearn.pipeline import Pipeline from sklearn.metrics import f1_score def main(): dataset = load_digits() X, y = dataset.data, dataset.target X = np.hstack([X, np.random.normal(size=(X.shape[0], X.shape[1]*2))]) X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.5, stratify=y, shuffle=True, random_state=0) sp = SelectPercentile() rfc = RandomForestClassifier(n_jobs=-1) pl = Pipeline([("sp", sp), ("rf", rfc)]) fig, ax = plt.subplots() ps = [10*x for x in range(1, 11)] for n_estimators in [50, 100, 150, 200]: f1s = [] for p in ps: pl.set_params(sp__percentile=p, rf__n_estimators=n_estimators) pl.fit(X_train, y_train) y_pred = pl.predict(X_test) f1s.append(f1_score(y_test, y_pred, average="macro")) ax.plot(ps, f1s, label="RF_n:" + str(n_estimators)) plt.legend() plt.savefig("result.png") if __name__ == "__main__": main()
結果です。

ほら、変数選択した方が結果が良い。一番良いのは30%のときなので、57個くらいの変数が使われたことになります。
考察
思考実験でも示した通り、理屈の上ではやったほうが良いのですが、これを実験で示そうとするとけっこう難しかったというのが率直な感想です。
上述の通り、ここに載せていないものも色々試したのですが、まず「明らかに無意味なノイズ特徴量」をわざと入れるくらいの人工的な条件でないと、特徴選択しない方が悪いという結果はほとんど得ることができませんでした。
ランダムフォレストを使っていると「変数選択しなくても(大真面目に変数選択してちゃんとチューニングしたときに得られる)最高性能に近い」ということはありがちで、「たとえ統計的に有意な関係がなくても、目的変数と何らかの形で関係がある(気がする)」、くらいの説明変数(特徴量)は入れておいてもそうそう悪さはしません。
適当に変数を取っているのでゴミがたくさん入っている、というようなシチュエーションでは威力を発揮することでしょう。
まとめ
わざとらしい感じはしますが、一応ランダムフォレストでも特徴選択した方が良いときはあり得る、ということは示せたのでよしとします。
*1:特に個体間の距離や内積に基づくものは変数選択しないとまずいでしょう
【python】sklearnのIterativeImputerで欠損値補完
注意:IterativeImputerは本記事の執筆時点(2019年11月)で実験的な実装とされており、最新の仕様等はこの記事の内容と異なる可能性があります。常にstable版の公式のドキュメントを確認してください。
公式のドキュメント
sklearn.impute.IterativeImputer — scikit-learn 0.21.3 documentation
はじめに
説明変数の欠損値補完には様々な方法があり、いろいろな研究がなされています。
直感的に思いつくのは、欠損部分は他の変数から回帰すれば良いという発想です。それを発展させて高級にすると、多重代入法というような手法になったりするらしいのですが、詳しくないので説明できません。どうせよく知らなくても機械学習モデルは構築できます。
scikit-learnではIterativeImputerなるモデルを「開発中」です。ここで開発中と書いたのは、公式ですら
Note This estimator is still experimental for now: the predictions and the API might change without any deprecation cycle.
という扱いになっているからです。「いつの間にか仕様が変わったり、最悪消えたりしてても文句言うなよ!」といったところでしょうか。それでも一応masterブランチにマージされていて、普通にsklearnをインストールすれば(2019年11月時点では)使えます。
ということで、便利そうなので紹介します。ただし、そこそこ癖があるので気をつけます。
使い方
まずリファレンスにも書いてあることですが、こいつを使うためには余計なものもimportする必要があります。
from sklearn.experimental import enable_iterative_imputer # 本当におまじない from sklearn.impute import IterativeImputer # 使うのこっち
使っているlintによっては「imported but unused」とか言われたりしますが、無視してください。
あとはハイパーパラメータをどうするかですが、あんまり凝ったことはしないのをおすすめします。と言いつつ、何も設定しないで使うのもおすすめしません。
重要なパラメータを重要な順番に並べてみます。
- estimator=None,
内部で回帰を行って補完するので、回帰モデルを渡せるという訳です。デフォルトはベイジアンリッジ(sklearn.linear_model.BayesianRidge)になります。なんでデフォルトがこれなのかは後述します。
- sample_posterior=False
そのままだと単一代入法、Trueにすると多重代入法になるのだと思いますが、いかんせん私が知識不足で、またドキュメントの記述がそっけないので細かいアルゴリズムまではよくわかりません。多重代入法の方がかっこよくて性能も良さそうに思えますが、実はその場合は
Estimator must support return_std in its predict method if set to True.
という制約があり、これは割ときつい制約です(対応しているモデルはほとんどありません)。デフォルトのBayesianRidgeはベイズ法なので予測の標準偏差が出せるということから選定されていると思われます。
- max_iter=10
- tol=0.001
特に説明は要らないと思います。なんか上手く行かないときはこの辺をいじる(増やしてみる)。
ということで、議論の余地があるのは多重代入法にするのか単一代入法にするのかです。多重代入法の場合、現実的なestimatorの選択肢はたぶんベイジアンリッジだけで、これは「ベイズでリッジだからつよつよ」と見せかけて基本的にただの線形モデルです(回帰式が一次式のやつ)。機械学習に突っ込むようなデータだとこれは微妙なことが多そうなので、多重代入法を諦めて単一代入法にすることで自由に好きな非線形回帰手法を使うということにした方が良いケースが多いと思います。
あとは何で回帰するか? ですが、ここの回帰モデルのパラメータチューニングのことなんて考えたくもないので、適当な設定でもそこそこ動く奴を使います。と言って真っ先に思いつくのはランダムフォレストですね。
以上をまとめると、だいたいこんな感じで定義することになると思います。
from sklearn.ensemble import RandomForestRegressor from sklearn.experimental import enable_iterative_imputer from sklearn.impute import IterativeImputer rf = RandomForestRegressor(n_estimators=500, n_jobs=-1) imp = IterativeImputer(estimator=rf)
他のパラメータは気になったら適当に弄ってください。大抵は弄らなくても大勢に影響しません。あと、ランダムフォレストはそこそこ遅いので、気になったら木を浅くして本数を減らすことで高速向きのチューニングにするか、他のものも試してみてください。
実験
SimpleImputerのときと同じことを試します。
【python】sklearnのSimpleImputerで欠損値補完をしてみる - 静かなる名辞
irisを欠損させて補完して予測精度を見ます。
import numpy as np from sklearn.datasets import load_iris from sklearn.neighbors import KNeighborsClassifier from sklearn.model_selection import train_test_split from sklearn.ensemble import RandomForestRegressor from sklearn.experimental import enable_iterative_imputer from sklearn.impute import IterativeImputer from sklearn.pipeline import Pipeline from sklearn.metrics import classification_report # 再現性確保 np.random.seed(0) # データ生成 dataset = load_iris() X_train, X_test, y_train, y_test = train_test_split( dataset.data, dataset.target, stratify=dataset.target, random_state=0) mask = ~np.random.randint(10, size=X_train.shape).astype(np.bool) X_train[mask] = np.nan # モデル定義 knn = KNeighborsClassifier() # 処理 def drop(): print("# drop") idx = ~(np.isnan(X_train).any(axis=1)) X_train_, y_train_ = X_train[idx], y_train[idx] knn.fit(X_train_, y_train_) y_pred = knn.predict(X_test) print(classification_report( y_test, y_pred, digits=3, target_names=dataset.target_names)) def impute(): print("# impute") rf = RandomForestRegressor( n_estimators=200, max_depth=3, n_jobs=-1) imp = IterativeImputer(estimator=rf, max_iter=20) pl = Pipeline([("imputer", imp), ("KNN", knn)]) pl.fit(X_train, y_train) y_pred = pl.predict(X_test) print(classification_report( y_test, y_pred, digits=3, target_names=dataset.target_names)) drop() impute()
# drop
precision recall f1-score support
setosa 1.000 1.000 1.000 13
versicolor 0.929 1.000 0.963 13
virginica 1.000 0.917 0.957 12
accuracy 0.974 38
macro avg 0.976 0.972 0.973 38
weighted avg 0.976 0.974 0.974 38
# impute
precision recall f1-score support
setosa 1.000 1.000 1.000 13
versicolor 1.000 1.000 1.000 13
virginica 1.000 1.000 1.000 12
accuracy 1.000 38
macro avg 1.000 1.000 1.000 38
weighted avg 1.000 1.000 1.000 38
単に欠損値のある行を学習データから落とすより良いわけです。まあ、irisのときは平均値補完でやっても同じ結果になりましたが。
データセットをwineにしてみます。
# drop
precision recall f1-score support
class_0 0.812 0.867 0.839 15
class_1 0.750 0.667 0.706 18
class_2 0.538 0.583 0.560 12
accuracy 0.711 45
macro avg 0.700 0.706 0.702 45
weighted avg 0.714 0.711 0.711 45
# impute
precision recall f1-score support
class_0 0.800 0.800 0.800 15
class_1 0.733 0.611 0.667 18
class_2 0.533 0.667 0.593 12
accuracy 0.689 45
macro avg 0.689 0.693 0.686 45
weighted avg 0.702 0.689 0.691 45
dropより良くなるかと思いましたが、そうでもないようです。平均値で補完するよりは良いみたいですが……。
考察
そもそも欠損値補完というアプローチは、
- 欠損値が多すぎるとそもそもまともに予測できないし、予測の悪さが大勢に影響を及ぼして全体のパフォーマンスを悪化させかねない
- 欠損値が少ないならdropしても大勢に影響はない
という矛盾を抱えている訳ですが、そこそこの欠損があるときにdropするよりちょっと良くなるかな? という可能性を追求するためにあるものだと思います。
まとめ
使うの面倒くさいしこの先どうなるかは読めないけど、使えるみたいです。ただし、できるからといってパフォーマンスの上で凄い効能があるとか、そういうものではないみたいです。
【python】sklearnのSimpleImputerで欠損値補完をしてみる
はじめに
欠損値補完(nanの処理)はだいたいpandasでやる人が多いですが、最近のscikit-learnはこの辺りの前処理に対するサポートも充実してきているので、平均値で補完する程度であればかえってscikit-learnでやった方が楽かもしれません。
ということで、sklearn.impute.SimpleImputerを使ってみようと思います。
sklearn.impute.SimpleImputer — scikit-learn 0.21.3 documentation
使い方
とても単純です。とりあえず、これを見てください。
import numpy as np from sklearn.datasets import load_iris from sklearn.impute import SimpleImputer # 再現性確保 np.random.seed(0) # irisでやることにする iris = load_iris() X = iris.data.copy() # 全体の約1割のデータをnanに置き換える mask = ~np.random.randint(10, size=X.shape).astype(np.bool) X[mask] = np.nan print(X[:10]) # 表示 # モデルの定義 imp = SimpleImputer() # トランスフォーム X_imputed = imp.fit_transform(X) print(X_imputed[:10]) # 表示
結果
[[5.1 nan 1.4 0.2] [4.9 3. 1.4 0.2] [4.7 3.2 1.3 0.2] [4.6 3.1 1.5 0.2] [5. 3.6 1.4 0.2] [5.4 3.9 1.7 0.4] [4.6 3.4 nan 0.3] [5. nan 1.5 0.2] [4.4 2.9 1.4 0.2] [4.9 3.1 nan 0.1]] [[5.1 3.06296296 1.4 0.2 ] [4.9 3. 1.4 0.2 ] [4.7 3.2 1.3 0.2 ] [4.6 3.1 1.5 0.2 ] [5. 3.6 1.4 0.2 ] [5.4 3.9 1.7 0.4 ] [4.6 3.4 3.8162963 0.3 ] [5. 3.06296296 1.5 0.2 ] [4.4 2.9 1.4 0.2 ] [4.9 3.1 3.8162963 0.1 ]]
ご覧の通り、埋まります。桁数が違うので場所がわかりやすいですね。
欠損値部分は列の平均値で埋められます。これはSimpleImputerのstrategyパラメータで変更できます(たとえばstrategy="median"とすれば中央値で補完してくれます)。
なお、sklearnの他のモデルと同様、補完に使われる値は「学習データから取得される」ことを覚えておきましょう。
>>> import numpy as np >>> from sklearn.impute import SimpleImputer >>> imp = SimpleImputer() >>> imp.fit([[100], [101],[99]]) SimpleImputer(add_indicator=False, copy=True, fill_value=None, missing_values=nan, strategy='mean', verbose=0) >>> imp.transform([[np.nan], [1], [2], [0]]) array([[100.], # ここが1になったりはしない [ 1.], [ 2.], [ 0.]])
こういう望ましい性質があるので、pandasでやるより機械学習向きです。カテゴリ変数のときにも書いたことですが。
効果のほどを見る
欠損値のあるデータを単にdropするのと、平均値補完とどちらが良いのでしょうか?
せっかくなのでirisの分類で試してみます。なお、面倒臭いので欠損値を作るのは学習データだけです。また、分類器は議論の余地が少ないKNNとします。
import numpy as np from sklearn.datasets import load_iris from sklearn.neighbors import KNeighborsClassifier from sklearn.model_selection import train_test_split from sklearn.impute import SimpleImputer from sklearn.pipeline import Pipeline from sklearn.metrics import classification_report # 再現性確保 np.random.seed(0) # データ生成 dataset = load_iris() X_train, X_test, y_train, y_test = train_test_split( dataset.data, dataset.target, stratify=dataset.target, random_state=0) mask = ~np.random.randint(10, size=X_train.shape).astype(np.bool) X_train[mask] = np.nan # モデル定義 knn = KNeighborsClassifier() # 処理 def drop(): print("# drop") idx = ~(np.isnan(X_train).any(axis=1)) X_train_, y_train_ = X_train[idx], y_train[idx] knn.fit(X_train_, y_train_) y_pred = knn.predict(X_test) print(classification_report( y_test, y_pred, digits=3, target_names=dataset.target_names)) def impute(): print("# impute") imp = SimpleImputer() pl = Pipeline([("imputer", imp), ("KNN", knn)]) pl.fit(X_train, y_train) y_pred = pl.predict(X_test) print(classification_report( y_test, y_pred, digits=3, target_names=dataset.target_names)) drop() impute()
結果
# drop
precision recall f1-score support
setosa 1.000 1.000 1.000 13
versicolor 0.929 1.000 0.963 13
virginica 1.000 0.917 0.957 12
accuracy 0.974 38
macro avg 0.976 0.972 0.973 38
weighted avg 0.976 0.974 0.974 38
# impute
precision recall f1-score support
setosa 1.000 1.000 1.000 13
versicolor 1.000 1.000 1.000 13
virginica 1.000 1.000 1.000 12
accuracy 1.000 38
macro avg 1.000 1.000 1.000 38
weighted avg 1.000 1.000 1.000 38
補完の方が良いのは確かですが、どちらも良すぎて議論しづらいので、データセットを難易度が少し高いwineにします。
# 中略 from sklearn.datasets import load_wine # 中略 dataset = load_wine()
結果
# drop
precision recall f1-score support
class_0 0.812 0.867 0.839 15
class_1 0.750 0.667 0.706 18
class_2 0.538 0.583 0.560 12
accuracy 0.711 45
macro avg 0.700 0.706 0.702 45
weighted avg 0.714 0.711 0.711 45
# impute
precision recall f1-score support
class_0 0.737 0.933 0.824 15
class_1 0.769 0.556 0.645 18
class_2 0.538 0.583 0.560 12
accuracy 0.689 45
macro avg 0.682 0.691 0.676 45
weighted avg 0.697 0.689 0.682 45
今度はdropした方が良いです。このように、どう処理するのが良いのかは割とケースバイケースです。たとえば欠損値の出現率を変えると勝ち負けが変わります。
どんなときでも躊躇なく平均値で補完すれば良いとは言えないのが、ちょっと嫌な所です。
まとめ
なにはともあれ、気楽に使えるものがある、というのは素晴らしいことです。
余談
実はもう少し高級な補完をしてくれるIterativeImputerなるモデルも存在しますが、
This estimator is still experimental for now: the predictions and the API might change without any deprecation cycle.
sklearn.impute.IterativeImputer — scikit-learn 0.21.3 documentation
という扱いなのと、軽く触ってみた感じそこそこ使いづらいので、今後紹介するかどうかは未定です。気が向いたら書こうと思います。
追記:けっきょく書きました。
www.haya-programming.com
mecab-pythonで品詞を見るときはfeature.splitしない方が速い
はじめに
mecab-pythonで形態素解析を行って何らかの処理をするとき、特定の品詞だけ取り出したいということがよくあります。
そういう目的で書かれたコードとして、よくこんなものを見たりすると思います。
import MeCab tagger = MeCab.Tagger() tagger.parse("") node = tagger.parseToNode("MeCabのテストをする") lst = [] while node: if "名詞" == node.feature.split(",")[0]: # ここが問題 lst.append(node.surface) node = node.next print(lst) # => ['MeCab', 'テスト']
それはそれで良いんですが、改めて冷静に考えてみると「str.splitはそこそこ負荷の高い処理なので、全形態素に対してやるのは問題にならないか?」という懸念が生じてきます。
ということで、他の方法がないのか考えて速度を比較してみようと思います。
やり方の検討
featureはIPA辞書を使っていれば、
蟻 名詞,一般,*,*,*,*,蟻,アリ,アリ
のように出てきます(タブのあとの部分がfeatureです)。
よって、これでもできます。
if "名詞," in node.feature:
この方法だと「名詞」という単語が出てくると若干問題があるのですが、実用上はほとんど問題ないでしょうし、回避策もその気になれば色々あるでしょう。
これと同じことは正規表現でもできます。正規表現エンジンでやった方が速いかもという淡い期待を抱いて、そちらも試すことにします。
実験
まず実験用データとして、wikipediaの「形態素解析」の記事の冒頭部分をコピペして、data.txtとして実行時カレントディレクトリに保存しておきました。文字数は445文字になりました。
その上で以下のコードを書いて実行しました。
import re import timeit import MeCab with open("data.txt") as f: text = f.read() tagger = MeCab.Tagger("") tagger.parse("") def f1(): """分割して条件式を作成 """ lst = [] node = tagger.parseToNode(text).next while node.next: feature = node.feature.split(",") if feature[0] == "名詞": lst.append(node.surface) node = node.next return lst def f2(): """カンマ区切りのfeatureに対してinで直接調べる """ lst = [] node = tagger.parseToNode(text).next while node.next: if "名詞," in node.feature: lst.append(node.surface) node = node.next return lst def f3(): """正規表現を使う """ r = re.compile(r"名詞,") lst = [] node = tagger.parseToNode(text).next while node.next: if r.match(node.feature): lst.append(node.surface) node = node.next return lst # 確認 print(f1() == f2() == f3()) # 計測 print(timeit.timeit(f1, number=1000)) print(timeit.timeit(f2, number=1000)) print(timeit.timeit(f3, number=1000))
結果は以下のようになりました。
True 1.841627002999303 1.6039621180025279 1.7953744690021267
案の定、inが一番速いです。正規表現はsplitするよりは速いものの、inには負けるようです。
解説
str.splitを行うと、
- splitの結果生じた各strオブジェクトの生成
- それを格納するlistオブジェクトの生成
という処理が行われ、新規にメモリ領域を確保するタイプの処理が走るため*1、とても遅くなります。ついでにいうと、参照カウンタも余計に走ります。
inの操作は値比較だけでできます。また、処理系の組み込み型の比較演算などは、多くの場合は極めて高度な最適化が施されていて、比較的に高速に実行できます。
まとめ
まあでも、これで期待される改善幅は1割とかそんなものなので、柔軟性と可読性を犠牲にしてまでやるかというと微妙かもしれません。
むしろ安直に書きたいときに使えます。
*1:処理系内部でバッファ領域はあると思いますが
【python】matplotlibのboxplotで外れ値を表示しないようにする
はじめに
matplotlibのboxplotを使うと簡単に箱ひげ図が描けます。ただし、デフォルト設定では外れ値が黒い円で表示されます。
どんなデータでも、サンプル数が多いと一定数の外れ値は出てしまいます。ただ、図を見る人は気にするところですし、外れ値がたくさんあると見た目にも悪いので、何らかの処置が必要です。
外れ値が描画されてしまうプログラムの例
import numpy as np import matplotlib.pyplot as plt np.random.seed(0) x = np.random.normal(size=3*10**3) # サンプル数に注目 plt.boxplot(x) plt.savefig("result1.png")

ということで、外れ値を表示させない方法を解説します。なお、この記事の内容は公式リファレンスに基づいています。
シンプルに表示させない
boxplotのキーワード引数のsymを使うと外れ値を「描画させない」設定が可能になります。具体的には、空文字列を指定します。
import numpy as np import matplotlib.pyplot as plt np.random.seed(0) x = np.random.normal(size=3*10**3) plt.boxplot(x, sym="") # 変更点 plt.savefig("result2a.png")

注意点としては、これはあくまでも外れ値の描画を行わないだけで、外れ値そのものはデフォルト通り計算されるということが挙げられます。つまり、本来の最大値・最小値よりひげが短くなります。
プロット上でだけ「消している」ということですね。
参考:
4-3. 外れ値検出のある箱ひげ図 | 統計学の時間 | 統計WEB
また、外れ値のマーカーに好きな記号を指定したりすることもできます。本来はこの用途で用いる引数です。
import numpy as np import matplotlib.pyplot as plt np.random.seed(0) x = np.random.normal(size=3*10**3) plt.boxplot(x, sym="+") # 変更点 plt.savefig("result2b.png")

使えるマーカーの一覧はこちらを参照。
matplotlib.markers — Matplotlib 3.1.1 documentation
外れ値の計算そのものをやめる
(外れ値も含めた)本来の最大値・最小値に基づいてひげを出す場合は、whis="range"を指定します。
そもそも外れ値の概念なしで箱ひげ図を描くというやり方になります。
import numpy as np import matplotlib.pyplot as plt np.random.seed(0) x = np.random.normal(size=3*10**3) plt.boxplot(x, whis="range") # 変更点 plt.savefig("result3.png")

この方が誤解を招く恐れがないので(つまり、外れ値検出をやっておいて外れ値をプロットしないという図は少しイレギュラーで伝わりづらい気がするので)、これでやるのがおすすめです。ただし、外れ値が少なくて、そんな極端に外れていないとき向きのやり方です(やたらひげが長いのも不格好ですから……)。
まとめ
二つやり方がありますが、意味が微妙に違うし、実際にそれぞれで異なった結果になるので注意してください。
matplotlibで図全体にタイトルを付けるにはsuptitleを使う
はじめに
matplotlibではよく一つの図の中に複数のグラフを描きます。そうすると全体に共通してタイトルを付けたくなるのですが、普通にやろうとしても個別のAxesに対して呼んでしまいがちです。
図全体に対してタイトルを付けるには、suptitleを使います。
使い方
以下のように使います。
import numpy as np import matplotlib.pyplot as plt # データ生成 x = np.arange(0, 10, 0.1) y1 = x ** 2 y2 = np.sin(x) # figure, axesの生成 fig, axes = plt.subplots(nrows=1, ncols=2) # 個別のAxesに対するプロットとタイトル付け axes[0].plot(x, y1) axes[0].set_title("y = x^2") axes[1].plot(x, y2) axes[1].set_title("y = sin(x)") # figure全体のタイトル fig.suptitle("graphs") # 保存 plt.savefig("result.png")

結果の図を見ればわかる通り、図の上に大きなタイトルが付いています。このように全体にタイトルを付けることができます。なお、今回はfigureのメソッドとして呼んでいますが、例のごとくpltからplt.suptitleという形でも呼べます。
今回は行数が少ないので大丈夫でしたが、たまにタイトル同士で被ったりしてグラフが不格好になることもあります。そういうときは適宜余白を調整してください。
matplotlibでAxesを真っ白にする(x軸とかy軸なんかを消して非表示にする)
はじめに
matplotlibでsubplotsを使って適当にグラフを並べるのはよくある処理だと思います。しかし、きれいに長方形で配置できないときもあります。
import numpy as np import matplotlib.pyplot as plt np.random.seed(0) data = [(np.random.randint(10, size=(10, )), np.random.randint(10, size=(10, ))) for x in range(5)] fig, axes = plt.subplots(nrows=2, ncols=3) for (x, y), ax in zip(data, axes.ravel()): ax.scatter(x, y) plt.savefig("fig1.png")

タイル状に作るので、場合によっては無駄なAxesが生成されます。これが邪魔なので消したいという場合、どうしたらいいのでしょうか?
解決策
ax.axis("off")みたいにすると消せます。
matplotlib.axes.Axes.axis — Matplotlib 3.1.0 documentation
あとはこれが呼ばれるようにすればオッケーです。やり方はいろいろありますが(それこそデータを1つずつ個別にプロットすれば簡単)、今回はforループとzipを使っているので、zip_longestで対応してみましょう(この辺は臨機応変に書いてください)。
from itertools import zip_longest import numpy as np import matplotlib.pyplot as plt np.random.seed(0) data = [(np.random.randint(10, size=(10, )), np.random.randint(10, size=(10, ))) for x in range(5)] fig, axes = plt.subplots(nrows=2, ncols=3) for xy, ax in zip_longest(data, axes.ravel()): if xy is None: ax.axis("off") continue x, y = xy ax.scatter(x, y) plt.savefig("fig2.png")

うまくいきました。右下がちゃんと真っ白になっています。
まとめ
私は長年これを知らなくて、画像編集で消したりしていたのですが、ax.axis("off")を覚えたので、これで今後はかっこいいグラフがスマートに描けます。
【python】機械学習でpandas.get_dummiesを使ってはいけない
はじめに
「pandasのget_dummiesでダミー変数が作れるぜ」という記事がとてもたくさんあって初心者を混乱させているのですが、これは「データ分析」には使えても「機械学習」には向きません。もう少し正確に言い換えると「訓練データからモデルを作り、未知のデータの予測を行うタスク」には使い勝手が悪いものです。
機械学習に使ってはいけないというのは大げさかもしれませんが、でも間違った使い方をよく見かけますし、こう言い切った方がぶっちゃけ良いと思います。
この記事では「こういうときにはget_dummies使うんじゃねえ!」ということと、どういう問題があるのかと、代替方法について書きます。
問題点
こんなデータを考えましょう。
>>> import pandas as pd >>> df = pd.DataFrame({"A":["hoge", "fuga"], "B":["a", "b"]}) >>> df A B 0 hoge a 1 fuga b >>> pd.get_dummies(df) A_fuga A_hoge B_a B_b 0 0 1 1 0 1 1 0 0 1
問題なさそうに見えますか?
でも、複数のデータに対して適用しようとするととたんに大問題が発生します。普通、kaggleのコンペとかならtrainとtestのデータはあるわけですよね。
>>> df_train = pd.DataFrame({"A":["hoge", "fuga"], "B":["a", "b"]})
>>> df_test = pd.DataFrame({"A":["hoge", "piyo"], "B":["a", "c"]})
>>> pd.get_dummies(df_train)
A_fuga A_hoge B_a B_b
0 0 1 1 0
1 1 0 0 1
>>> pd.get_dummies(df_test)
A_hoge A_piyo B_a B_c
0 1 0 1 0
1 0 1 0 1
shapeは同じ。だけど、各カラムの意味するものは異なっています。一致しているのはB_aだけという惨状です。
ユニークな要素は6つあるので、下のようになればまずまずの結果と言っていいかもしれません。
# trainに対して A_fuga A_hoge A_piyo B_a B_b B_c 0 0 1 0 1 0 0 1 1 0 0 0 1 0 # testに対して A_fuga A_hoge A_piyo B_a B_b B_c 0 0 1 0 1 0 0 1 0 0 1 0 0 1
実際は学習データに含まれない値なんて落ちてくれて構わない(逆に落ちないと厄介)ので、理想的な結果はこうでしょうか。
# trainに対して A_fuga A_hoge B_a B_b 0 0 1 1 0 1 1 0 0 1 # testに対して A_fuga A_hoge B_a B_b 0 0 1 1 0 1 0 0 0 0
ドキュメントを軽く読んでいろいろ試した感じ、これをget_dummiesで得るのは無理っぽいです。つかえねー。
(私が見落としているだけかもしれないので、「できるよ」という人はコメントで教えて下さい。確認した上で記事に反映させていただきます。)
(↑さっそくコメントを頂いて、追記させていただきました。この章の末尾を御覧ください。)
こういう問題があるので、get_dummiesはダメと言っています。
なお、先に示した6列のデータなら、pandas.concatしてから変換すれば得ることができます。
>>> ret = pd.get_dummies(pd.concat([df_train, df_test])) A_fuga A_hoge A_piyo B_a B_b B_c 0 0 1 0 1 0 0 1 1 0 0 0 1 0 0 0 1 0 1 0 0 1 0 0 1 0 0 1 >>> ret = pd.get_dummies(pd.concat([df_train, df_test])) >>> X_train, X_test = ret.iloc[:2], ret.iloc[2:] >>> X_train A_fuga A_hoge A_piyo B_a B_b B_c 0 0 1 0 1 0 0 1 1 0 0 0 1 0 >>> X_test A_fuga A_hoge A_piyo B_a B_b B_c 0 0 1 0 1 0 0 1 0 0 1 0 0 1
これをやっているコードも見かけたことがありますが、「予測モデル側で学習データぜんぶ持っておくの?」ということを考えると現実的なソリューションではないでしょう。おすすめしません。
追記
列をpandas.Categorical型とすれば、明示的にカテゴリを指定することで変換が可能なようです。
df_train = pd.DataFrame({"A": ["hoge", "fuga"], "B": ["a", "b"]}) df_test = pd.DataFrame({"A": ["hoge", "piyo"], "B": ["a", "c"]}) df_test["A"] = pd.Categorical(df_test["A"], categories=["hoge", "fuga"]) df_test["B"] = pd.Categorical(df_test["B"], categories=["a", "b"]) pd.get_dummies(df_test)テナジマ様コメントより
scikit-learnでやるのと比べて記述は増えますが、pandasの枠組みの中で取り扱うこと自体は可能なようです。
代替する方法
sklearnのOneHotEncoderで変換すれば一発です。
sklearn.preprocessing.OneHotEncoder — scikit-learn 0.21.3 documentation
>>> from sklearn.preprocessing import OneHotEncoder >>> ohe = OneHotEncoder(handle_unknown="ignore", sparse=False) >>> ohe.fit(df_train) OneHotEncoder(categorical_features=None, categories=None, drop=None, dtype=<class 'numpy.float64'>, handle_unknown='ignore', n_values=None, sparse=False) >>> ohe.transform(df_train) array([[0., 1., 1., 0.], [1., 0., 0., 1.]]) >>> ohe.transform(df_test) array([[0., 1., 1., 0.], [0., 0., 0., 0.]])
一撃で理想的な結果を得られています。scikit-learnは偉大ですね。
pandasなんて最初から要らなかった。
これはscikit-learnのモデルなので、Pipelineなどと組み合わせて使うのにも親和性が高いです。というか、そのように使ってください(Pipelineにすることで、transformするべきところでfit_transformするといった凡ミスを防げます)。
使い方についてはこっちの記事も参照してください。
【python】sklearnでのカテゴリ変数の取り扱いまとめ LabelEncoder, OneHotEncoderなど - 静かなる名辞
ColumnTransformerと組み合わせると使い方の幅が広がります。カテゴリ変数だけ投げて数値変数はそのまま通すといった処理が可能になります。
scikit-learnのColumnTransformerを使ってみる - 静かなる名辞
あとこれは完全に余談ですが、その気になればnanもsklearnの中で落とせます。前処理からすべてscikit-learnの枠組みの中で書けるので、pandasの出る幕はCSVの読み込みと探索的データ分析でやる各種プロットとか以外にはないと言っても過言ではないでしょう。
5.4. Imputation of missing values — scikit-learn 0.21.3 documentation
……え、結果がpandasのDataFrameになってないのがいやだって? そしたら、結果を改めてDataFrameに変換すればいいんじゃないでしょうか。こんな感じですね。
>>> pd.DataFrame(ohe.transform(df_test), columns=[c + "_" + x for lst, c in zip(ohe.categories_, "AB") for x in lst]) A_fuga A_hoge B_a B_b 0 0.0 1.0 1.0 0.0 1 0.0 0.0 0.0 0.0
まとめ
pandasは基本的にこういう用途には向いていないので、安易に使わないほうが良いという話です。機械学習ライブラリとして枠組みを整備してくれているscikit-learnは偉大なツールなので、積極的にこっちを活用していけばいいと思います。
【python】クラスを関数として使う
はじめに
クラスはcallされたら自分のクラスのインスタンスを返さないといけないと思っていませんか? 一般論としてはその通りなのですが、Pythonではそうしないメカニズムも用意されています。
__new__を使えば割となんでもできます。もっとも、実用的な用途は相当限られるでしょう。
原理
こんな感じ。
クラス
クラスは呼び出し可能です。そのオブジェクトは通常、そのクラスの新たなインスタンスのファクトリとして振舞いますが、 __new__() をオーバーライドして、バリエーションを持たせることもできます。クラス cls の新しいインスタンスを作るために呼び出されます。
なんのこっちゃと思うかもしれませんが、
- Hogeがcallされる(コード的にはHoge(*args, **kwargs))
- Hoge.__new__(Hoge, *args, **kwargs)がcallされる。返り値はHogeのインスタンス(を適切に生成して返すのが__new__の役割ということ)で、この段階でselfができる
- Hoge.__init__がcallされる(いつもの)
という流れで処理されている訳ですね。
やってみる
以上を理解したら、自由自在にクラスを関数として使えます。
class add: def __new__(cls, a, b): return a + b print(add(1, 2)) # => 3
意外と単純です。
なお、目ざとい人は「あれ、__init__の引数フォーマットでエラーにならない?」と思うかもしれませんが、上述のドキュメントにも書いてある通り
__new__() が cls のインスタンスを返さない場合、インスタンスの __init__() メソッドは呼び出されません。
という仕様があり、上記の記述を可能にしています。
実用的(?)な用途
別にわざわざこんなことをする必要はないのですが、使いみちがまったくないわけではありません。
引数によってインスタンスを返すときとそうでないときがあったりする
コンストラクタに引数が与えられたかどうかによって返り値の型が変わるパターン。
class Hoge: def __new__(cls, a=None): if a is None: return "this is not Hoge's instance, is a str" else: self = super().__new__(cls) return self def __init__(self, a=None): self.a = a def __repr__(self): return "Hoge(a={})".format(self.a) h1 = Hoge() h2 = Hoge(42) print(h1) # => this is not Hoge's instance, is a str print(h2) # => Hoge(a=42)
面白い使いみちがあるかもしれません。かえって厄介かもしれません。
デコレータを書くのに使う
デコレータをクラスで作るというテクニックがあります。
参考:
【python】クラスでデコレータ! - 静かなる名辞
で、このときに
@deco def f(args): ... @deco(options) def f(args): ...
の両方に対応させたいケースがあります。面倒くさいですね。
こういうときは__new__で切り分ける実装になるのだと思いますが、書こうとしたら相当ややこしかったのでコードは割愛させてください。
まとめ
知れば知るほど奥が深い動的言語の世界。
クラスが自分のインスタンスを返さなくても良いということは知らなかった。
【python】scikit-learnで大規模疎行列を扱うときのTips
はじめに
自然言語処理などで大規模疎行列を扱うことがあります。一昔前はNLPといえばこれでした(最近は低次元密行列で表現することのほうが多いですが)。
疎行列はその特性をうまく生かして扱うとパフォーマンス上のメリットが得られる反面、うかつにdenseな表現に展開してしまうと効率が悪くなって激遅になったり、あっさりメモリから溢れたりします。
scikit-learnでやる場合、うっかり使うと自動的にdenseな表現に展開されてしまう、という事故が起こりがちで、要するに使えるモデルに制約があり、注意が必要です。その辺の基本的なことをまとめておきます。
目次
疎行列ってなに?
まず、Pythonで疎行列といえばscipy.sparse.csr_matrixなどのことです。
scipy.sparse.csr_matrix — SciPy v1.4.1 Reference Guide
内部の構造の詳細などは、こちらの記事が参考になります。
重要なのは、まずこの方式にすると「メモリ効率がいい」ということです。これは単純に嬉しいですし、CPUキャッシュのことを考えても、パフォーマンス上大きなメリットがあります。また、0の要素は飛ばして探索できるので、うまく使うと効率も良くなります。
大規模疎行列を相手にするときは、できるだけ疎行列のまま取り扱うことが重要になります。
特徴抽出する
自然言語処理系のタスクだと、CountVectorizerやTfidfVectorizerが使えます。どちらもデフォルトでcsr_matrixを返してくれるので、素直に使えば疎行列が出てきます。
sklearn.feature_extraction.text.CountVectorizer — scikit-learn 0.22.1 documentation
sklearn.feature_extraction.text.TfidfVectorizer — scikit-learn 0.22.1 documentation
もう少し幅広いタスクで使いたい場合は、DictVectrizerが便利でしょう。こちらもデフォルトではsparseな表現を返します(オプションでnumpy配列を返すようにすることも可能)。
sklearn.feature_extraction.DictVectorizer — scikit-learn 0.22.1 documentation
特徴選択する
特徴抽出したあと素直に使うとだいたい変数が多すぎて使いづらいので、普通は変数選択をすると思います。sklearn.feature_selectionのものなら、これはだいたいデフォルトで疎行列のままの取り扱いに対応しています。
1.13. Feature selection — scikit-learn 0.22.1 documentation
疎行列としてinputすれば疎行列で出てきます。速度もそうした方が速いです。
標準化する
StandardScalerで標準化する場合は、with_mean=Falseを指定してください。これは平均0にしない標準化です。標準化の式の分子で平均を引かないものです。
それだけで疎行列型のまま標準化することができます。
This scaler can also be applied to sparse CSR or CSC matrices by passing with_mean=False to avoid breaking the sparsity structure of the data.
sklearn.preprocessing.StandardScaler — scikit-learn 0.22.1 documentation
次元削減する
Truncated SVDという素晴らしい手法があり、実装上も疎行列に対応しているので、こちらを使ってください。逆に、これ以外の選択肢は(おそらく)ありません。
sklearn.decomposition.TruncatedSVD — scikit-learn 0.22.1 documentation
ただし、次元削減した方が良いのか、しない方が良いのかはなんとも言えません。次元削減は行わないで、疎行列型のまま後段のモデルに突っ込むという選択もあるからです。ぶっちゃけ性能は大して変わらないし、次元削減に時間がかかるのと大規模密行列になってしまうぶんだけ遅くなるかもしれない……という微妙な性質があります。
それでも、次元削減が必要ならやればできます。
その他の各種transformerを使う
transformの返り値がsparse matrixになるかどうかを確認してください。油断しているとnumpy配列に変換されます。
リファレンスからある程度は読み取れますが、わからないことも多いので、一度動かして確かめた方が良いと思います。
分類や回帰など、予測に使う
Truncated SVDで次元削減をした場合は勝手にnumpy配列になっているので、どんなモデルにも入力できます(実用的な速度と性能が両立できるかは別)。
csr_matrixのまま突っ込む場合は、そのまま入力できるestimatorとできないestimatorがあるので、注意が必要です。これを確認するには、リファレンスのfitメソッドのパラメータを見ます。
たとえばRandomForestClassifierだと、
X : array-like or sparse matrix of shape = [n_samples, n_features]
3.2.4.3.1. sklearn.ensemble.RandomForestClassifier — scikit-learn 0.22.1 documentation
という記述があり、sparse matrixと書いてあるのが「疎行列型でも受け付けて、適切に取り扱ってあげますよ」という意味です。一方、たとえばLinearDiscriminantAnalysisだと(あまり使う人もいないと思いますが)、
X : array-like, shape (n_samples, n_features)
sklearn.discriminant_analysis.LinearDiscriminantAnalysis — scikit-learn 0.22.1 documentation
と書いてあります。array-likeのときは、渡せば動くけど、内部的にはdenseな表現(numpy配列)に変換されてしまう公算が大きいです。でもけっきょくのところはよくわからないので、実際に入れて動くかどうか試してみた方が良いでしょう。
他に実例は省略しますがnumpy arrayと書いてあるときも(たぶん)あり、この場合はたぶんsparse matrixだとエラーを吐きます。
実際に動かしてみないと挙動がわからないこともままあるので、突っ込んでみてエラーが出ないか、メモリ消費が異常に膨れ上がらないかを確認しながら作業した方が良いと思います。
以下は疎行列型でも行ける(と思う)代表的なestimatorのリストです。
分類器
- sklearn.ensemble.RandomForestClassifier
- sklearn.svm.SVC
- sklearn.svm.LinearSCV
- sklearn.naive_bayes.MultinomialNB
非負のみ。文書分類向き
- sklearn.linear_model.LogisticRegression
回帰モデル
- sklearn.ensemble.RandomForestRegressor
- sklearn.svm.SVR
- sklearn.linear_model.ElasticNet
代表的なものはだいたい対応しているけど、たまに使えないのもあるという感じです。
実際にやってみる
20newsgroupsの文書ベクトルを返してくれるものがあるので、それでやります。
Classes 20
Samples total 18846
Dimensionality 130107
Features realsklearn.datasets.fetch_20newsgroups_vectorized — scikit-learn 0.22.1 documentation
sklearnのfetch_20newsgroups_vectorizedでベクトル化された20 newsgroupsを試す - 静かなる名辞
ご覧の通りでかいので、ナイーブにnumpy配列に変換して扱おうとすると苦労します。前にやったときは密行列に変換しようとしていろいろ苦労していましたが、疎行列型のままやった方がシンプルです。
もちろん通例通り、Pipelineを使ってモデルを組み合わせます。
【python】sklearnのPipelineを使うとできること - 静かなる名辞
from sklearn.datasets import fetch_20newsgroups_vectorized from sklearn.feature_selection import SelectKBest from sklearn.decomposition import TruncatedSVD from sklearn.svm import LinearSVC from sklearn.pipeline import Pipeline from sklearn.metrics import classification_report def main(): train = fetch_20newsgroups_vectorized(subset="train") test = fetch_20newsgroups_vectorized(subset="test") X_train, y_train = train.data, train.target X_test, y_test = test.data, test.target target_names = train.target_names skb = SelectKBest(k=5000) tsvd = TruncatedSVD(n_components=1000) svm = LinearSVC() clf = Pipeline([("skb", skb), ("tsvd", tsvd), ("svm", svm)]) clf.fit(X_train, y_train) y_pred = clf.predict(X_test) print(classification_report( y_test, y_pred, target_names=target_names)) if __name__ == "__main__": main()
実行したところ、1分くらいで処理が完了しました。パフォーマンスのよさが伺えます。
結果。
precision recall f1-score support
alt.atheism 0.70 0.68 0.69 319
comp.graphics 0.70 0.71 0.70 389
comp.os.ms-windows.misc 0.71 0.71 0.71 394
comp.sys.ibm.pc.hardware 0.66 0.64 0.65 392
comp.sys.mac.hardware 0.76 0.76 0.76 385
comp.windows.x 0.79 0.72 0.75 395
misc.forsale 0.81 0.87 0.84 390
rec.autos 0.83 0.83 0.83 396
rec.motorcycles 0.91 0.90 0.90 398
rec.sport.baseball 0.84 0.89 0.87 397
rec.sport.hockey 0.92 0.95 0.94 399
sci.crypt 0.89 0.88 0.89 396
sci.electronics 0.66 0.65 0.66 393
sci.med 0.81 0.78 0.79 396
sci.space 0.86 0.87 0.86 394
soc.religion.christian 0.76 0.91 0.83 398
talk.politics.guns 0.68 0.88 0.77 364
talk.politics.mideast 0.91 0.81 0.85 376
talk.politics.misc 0.71 0.54 0.62 310
talk.religion.misc 0.61 0.41 0.49 251
accuracy 0.78 7532
macro avg 0.78 0.77 0.77 7532
weighted avg 0.78 0.78 0.78 7532
今回は性能を重視していないのでこの程度です。このタスクだとできるだけ次元を維持したまま(疎行列型のまま)ナイーブベイズに入れたほうが速くて性能が出るという知見を以前に得ています。その場合は0.83くらいまで出ています。
まとめ
うまく疎行列型配列を使うと数桁くらい時間を節約できます。ぜひ活用してみてください。
こちらの記事もおすすめです。
scikit-learnのモデルに疎行列(csr_matrix)を渡したときの速度 - 静かなる名辞
【python】itertools.chainを使って複数のiterableを一つにまとめる
概要
複数のiterable(リストとか)を結合させてループさせたいときがあります。
>>> lst1 = [1, 2, 3] >>> lst2 = [4, 5, 6] >>> # 1, 2, 3, 4, 5, 6というループをやりたい
連結すればできたりしますが、余計なメモリを確保するのでスマートではないし、パフォーマンスが気になります。
>>> for x in lst1 + lst2: ... print(x) ... 1 2 3 4 5 6
というマニアックなお悩みを解決してくれるのがitertools.chainです。
使い方
リファレンスを見ると使い方が書いてあります。
itertools --- 効率的なループ実行のためのイテレータ生成関数 — Python 3.7.4 ドキュメント
まあまったく難しいことはなく、
>>> from itertools import chain >>> for x in chain(lst1, lst2): ... print(x) ... 1 2 3 4 5 6
たったこれだけで使えます。単純ですね。
速度を測る
実際にパフォーマンスがいいのかどうか試してみましょう。
import timeit from itertools import chain def f1(a, b): for x in a + b: pass def f2(a, b): for x in chain(a, b): pass def main(): for n in [10, 100, 1000, 10000]: print(n) a, b = list(range(n)), list(range(n, 2*n)) t1 = timeit.timeit(lambda : f1(a, b), number=1000) t2 = timeit.timeit(lambda : f2(a, b), number=1000) print("{0:.6f}\n{1:.6f}".format(t1, t2)) if __name__ == "__main__": main() """ => 10 0.000657 0.000822 100 0.005128 0.004024 1000 0.032347 0.025703 10000 0.249460 0.232873 """
なんかどっちでも良いような気が……論理的にはchainを使ったほうがスマートですが、
- リストの結合と値の取り出しは速い
- chainはかえって遅い
というあたりが本質かな、という気がします。
素のリストとchainの速度も比較してみる。
import timeit from itertools import chain def f1(lst): for x in lst: pass def f2(lst): for x in chain(lst): pass def main(): lst = list(range(10**4)) t1 = timeit.timeit(lambda : f1(lst), number=1000) t2 = timeit.timeit(lambda : f2(lst), number=1000) print("{0:.6f}\n{1:.6f}".format(t1, t2)) # 0.092083 # 0.130389 if __name__ == "__main__": main()
かえって遅い可能性もあるということです。
諦めて二重forで書く
これで良いんじゃ……
import timeit from itertools import chain def f1(a, b): for x in a + b: pass def f2(a, b): for x in chain(a, b): pass def f3(a, b): for lst in [a, b]: for x in lst: pass def main(): for n in [10, 100, 1000, 10000]: print(n) a, b = list(range(n)), list(range(n, 2*n)) t1 = timeit.timeit(lambda : f1(a, b), number=1000) t2 = timeit.timeit(lambda : f2(a, b), number=1000) t3 = timeit.timeit(lambda : f3(a, b), number=1000) print("{0:.6f}\n{1:.6f}\n{2:.6f}".format(t1, t2, t3)) if __name__ == "__main__": main() """ => 10 0.000675 0.000837 0.000726 100 0.003831 0.003594 0.002471 1000 0.033191 0.025650 0.016758 10000 0.279039 0.225997 0.163289 """
こんなのが一番速いです。無理に一つにまとめない方がパフォーマンス上良い、というしょうもない結論に。
まとめ
なんかchainするとパフォーマンス上は微妙な気もしますが、使うと連結よりスマートな感じのコードになるので、好みで使うと良いでしょう。
【python】namedtupleはすごい(きもちわるい)
概要
namedtupleは存在は知ってたけど、使い方を知ったら「すげえ(きめえ)」という感想にしかならなかったので、記事に書きました*1。
namedtupleはクラスではない
普通は、namedtupleというクラスがあると思いませんか? さにあらず、関数です。
>>> from collections import namedtuple >>> namedtuple <function namedtuple at 0x7f8c288800d0>
お、おう。使い方を調べます。
名前付きフィールドを持つタプルのサブクラスを作成するファクトリ関数
>>> # Basic example >>> Point = namedtuple('Point', ['x', 'y']) >>> p = Point(11, y=22) # instantiate with positional or keyword arguments >>> p[0] + p[1] # indexable like the plain tuple (11, 22) 33 >>> x, y = p # unpack like a regular tuple >>> x, y (11, 22) >>> p.x + p.y # fields also accessible by name 33 >>> p # readable __repr__ with a name=value style Point(x=11, y=22)
あー、そういうものだったの。
第一引数はクラス名、第二引数は「属性」の名前ですね。はい。こいつはクラスを返します。
大切なことなのでもう一度書きます。namedtupleはクラスを生成して返す関数です。
動的言語の面目躍如という感じ……。
一応タプルらしい
タプルらしく、中身を変えられないし、hashableです。
>>> Hoge = namedtuple("Hoge", "a b") # 空白orカンマ区切り文字列で属性リストにできる >>> h = Hoge(a=1, b=2) >>> h Hoge(a=1, b=2) >>> h[0] = 2 Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: 'Hoge' object does not support item assignment >>> h.a = 2 Traceback (most recent call last): File "<stdin>", line 1, in <module> AttributeError: can't set attribute >>> {h} {Hoge(a=1, b=2)}
だいたい期待する性質は持っていると言っていいでしょう。いや、気持ち悪いですが。
でもしょせんただのクラスなので
継承して好きなメソッドを定義したりできます。すごい!
>>> class Hoge2(Hoge): ... def product_of_a_and_b(self): ... return self.a * self.b ... >>> h2 = Hoge2(a=10, b=100) >>> h2.product_of_a_and_b() 1000
元のクラスが要らなければ、こういう書き方もあります。上のドキュメントに紹介されている例です。
>>> class Point(namedtuple('Point', ['x', 'y'])): ... __slots__ = () ... @property ... def hypot(self): ... return (self.x ** 2 + self.y ** 2) ** 0.5 ... def __str__(self): ... return 'Point: x=%6.3f y=%6.3f hypot=%6.3f' % (self.x, self.y, self.hypot) >>> for p in Point(3, 4), Point(14, 5/7): ... print(p) Point: x= 3.000 y= 4.000 hypot= 5.000 Point: x=14.000 y= 0.714 hypot=14.018
理解はできるけど、すごく○○です……。
まとめ
こういうものだとは知らなかったのと、発想が斜め上を行っているのと、いろいろな意味で驚きました。
クラス定義のコンストラクタ(__init__)の記述を省略したりするのにも使えるそうです。言われるとなるほどと思いますが、pythonはほんと奥が深いですね……。
*1:そんなことも知らないでPython書いてたのかよって感じですが